Page 1

生命经纬 www.biox.cn
Vector NTI7.0 使用手册
翻译者:宋厚辉(浙江大学)
一、前言(Introduction)...............................................................................................................1
二、Chapter 1...................................................................................................................................1
三、Chapter 2.................................................................................................................................10
四、Chapter 3.................................................................................................................................16
五、Chapter 4.................................................................................................................................20
一、前言(Introduction)
1. 程序附带的数据库(Vector NTI database)包括:DNA/RNA、蛋白质、内切酶、寡核苷
酸、凝胶 mark。此外程序还提供数据库开发(Database Explorer)功能,用户可以自己
修改、添加、拷贝感兴趣的各类数据库。
2. 创建新分子(有四种方法)
A. 用
B. 手工粘帖,然后保存到数据库中
C. 从其他分子、接头、载体中剪切、拼接构键
D. 从 DNA 或 RNA 分子的编码区翻译成蛋白质
3. 关于新分子的序列特征图谱:利用 GenBank/GenPept, EMBL/SWISS-PROT or FASTA 等
格式输入的分子都能显示出序列和结构图,但自己手工粘帖的没有,需要自己编辑
GenBank/GenPept, EMBL/SWISS-PRO T and FAST A、ASCII 等格式输入 DNA
或氨基酸。
二、Chapter 1
Tutorial: Display Windows(显示窗口)
目的:创建显示窗口,并对图、序列和文本进行操作
1、登录 Vector NTI
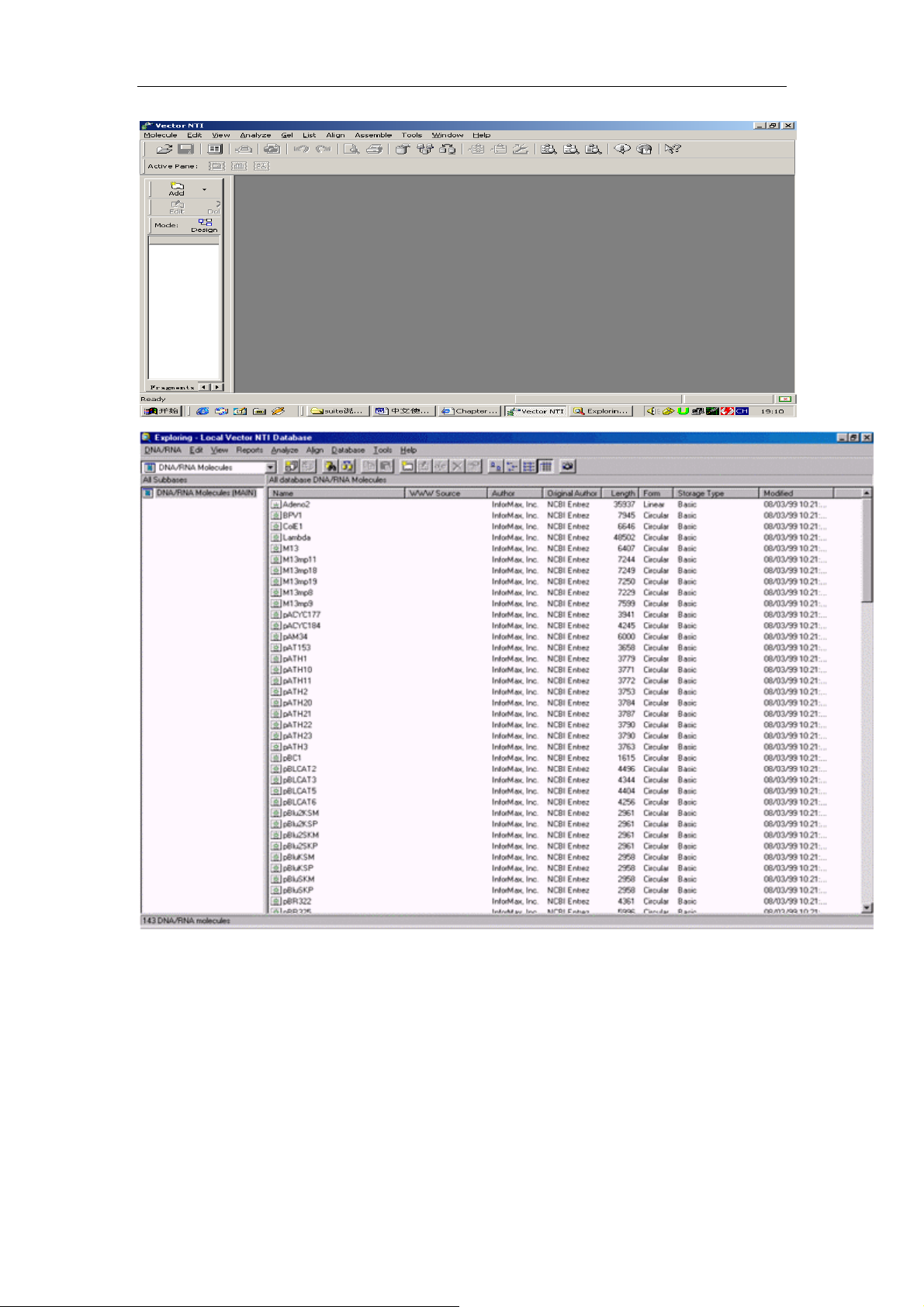
安装后首次登录,系统将提示是否允许填充空库,点 OK。这样 DNA molecules, proteins,
enzymes, oligos, and gel markers 将组成 NTI 的数据库。并出现下列两个窗口。
2、观察出现的 Vector NTI 工作窗口 和 Database Explorer 窗口
1
Page 2
生命经纬 www.biox.cn
上面的第一个窗口为工作窗口,由菜单栏和工具条两栏,移动鼠标到工具栏任意选项处,鼠
标自动显示每个工具条的功能。
第二个窗口为 exlporing——local vector NTI database,显示的是上次打开的 DNA/RNA 或蛋白
分子。
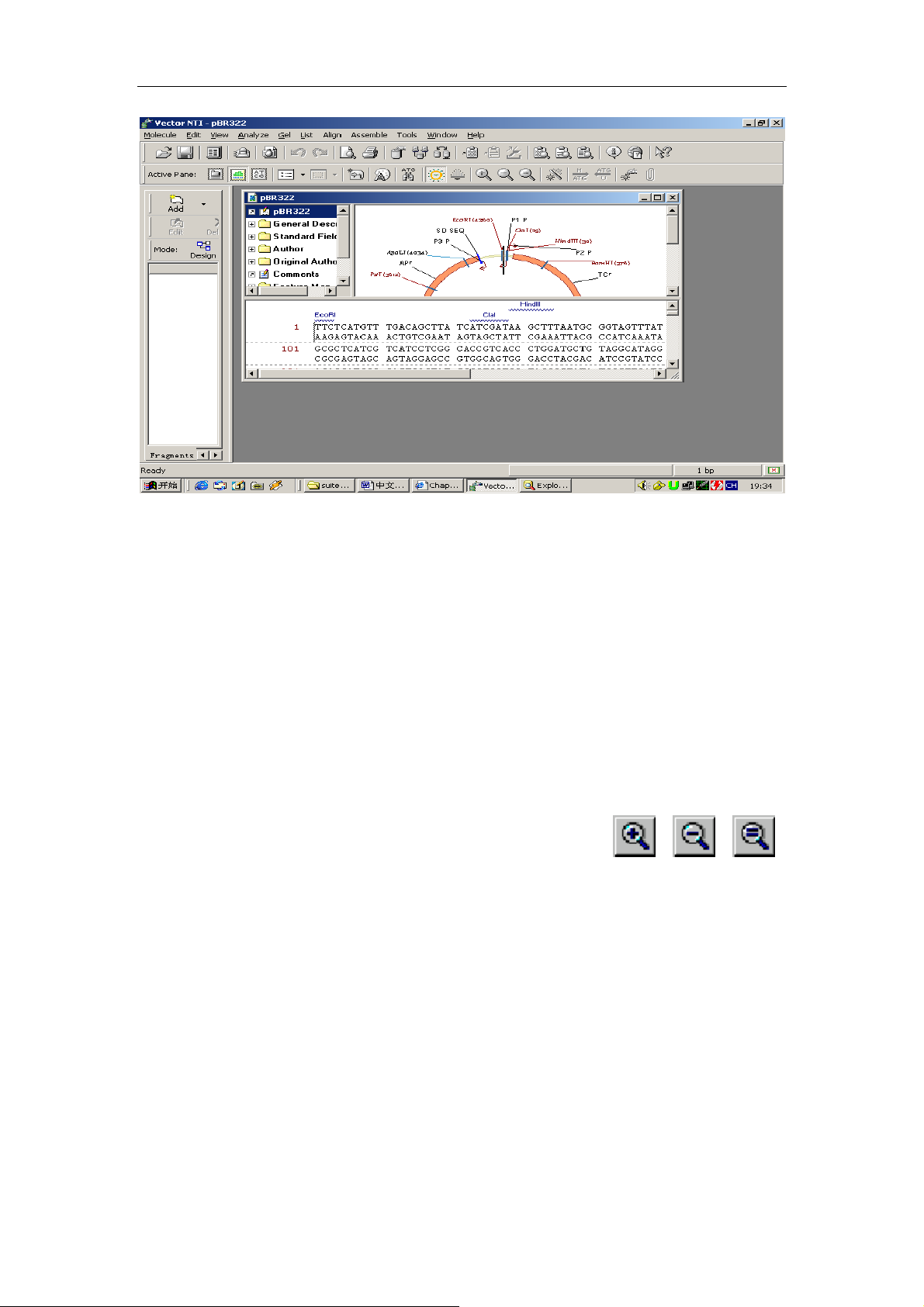
3、Create a Display Window for pBR322
激活 exlporing——local vector NTI database 窗口中的 DNA/RNA Molecules (MAIN) 数据库,
找到 pBR322 分子并双击打开。显示如下窗口:
2
Page 3
生命经纬 www.biox.cn
4、观察 pBR322 显示窗口
上面的窗口由文本区(对分子信息的文字描述,双击文件夹可以看到)、图形区(标住限制
性内切酶位点等)和序列区(全序列及酶切位点)三个部分组成。
5、显示窗口的管理(通过拖拉标尺,改变窗口、或每个显示区的相对大小)
6、转换 pBR322’s 图形区:在工具栏左边的 active pane 右侧有三个按
钮,用鼠标点当中那个(graphics pane)
7、 pBR322’s 结构图进行操作:用户可以尝试点击
、 、 ,
此时图形的大小会发生变化。选区设定:菜单 Edit——>set selection,
输入 100bp–1000bp,然后 OK.可以看到选取在图形中用扇型框圈了起
来.将鼠标移到选取的 5’端,可以看到
,通过拖拉可以延长或缩短选区
范围,同样在 3’端也能做到。如果用户一次只想移动一个碱基用直接拖拉
就可能不方便,假如用户想在 5’端移动一个碱基,首先将鼠标放到
处,
然后按住 shift 和键盘右侧的——>或<——箭头,则按一次箭头移动一
个碱基距离。如果一次想移动 10 个碱基,则同时按住 shift+ctrl+箭头。
如果用户只是粗略选择,可以直接将鼠标移到图中,用十字花拖动。将
鼠标移到图的 TCr 处,此时鼠标箭头变成
,并显示 TCr 代表的含义,
如果用户此时按下鼠标,则 TCr 的编码区将被选中。
3
Page 4
生命经纬 www.biox.cn
8、检查 pBR322’s nucleotide 序列
移动标尺,尽可能将更多的 PBR322 序列显示出来,并点击序列中任何一处。
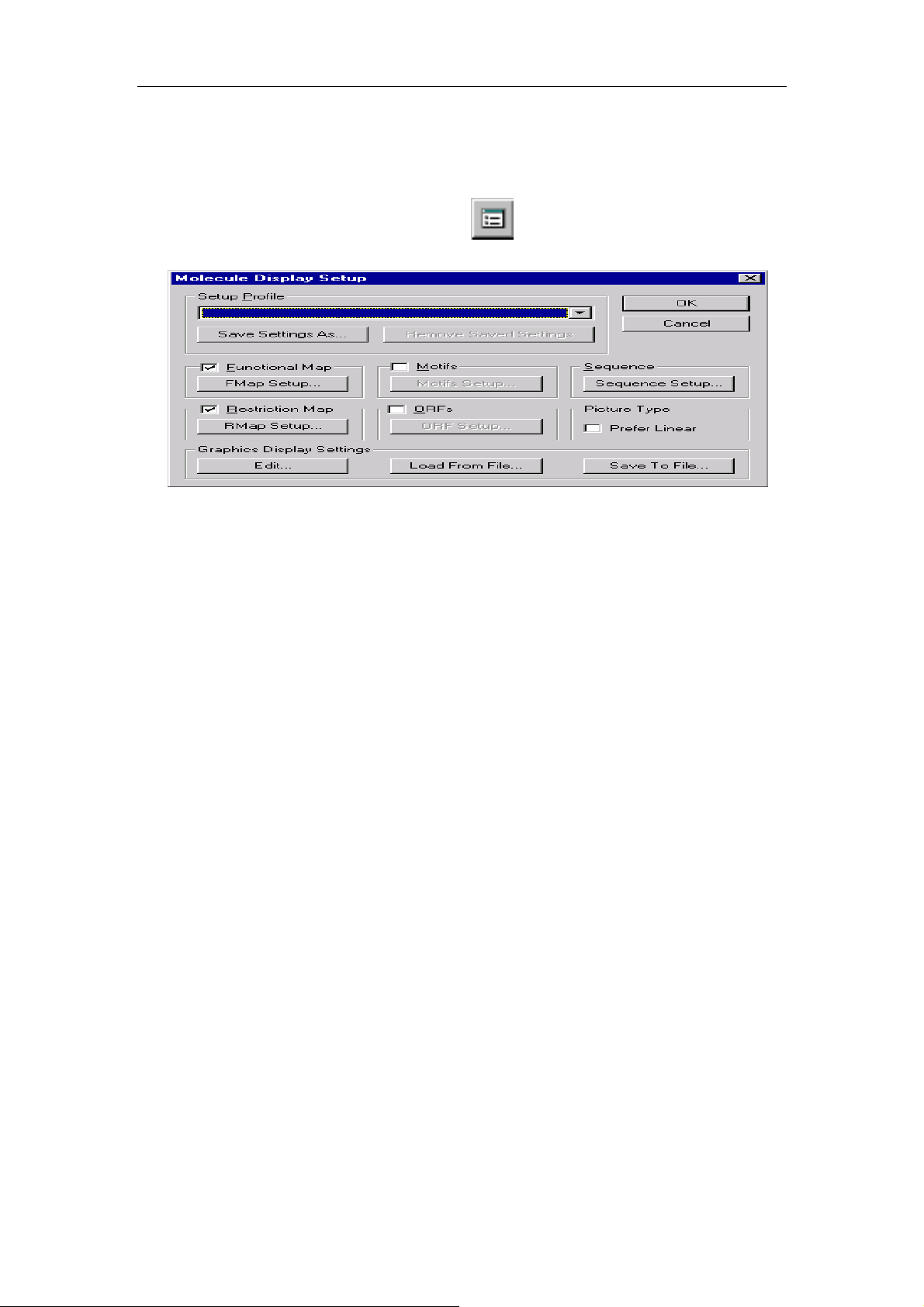
现在对序列显示的样式进行设定:点击
(Display Setup)按钮,显示
molecular display setup 对话框:
用户可以对序列的颜色、大小、10 个一组显示还是 15 个一组(默认是 10 个
碱基一组),结构图的颜色、限制酶切图谱(注意刚开始显示的 PBR322 上限
制酶并不多)、ORF、Motif 等进行设置。现在我们打算把序列字体的颜色由黑
色变成绿色,显示全部 PBR322 的酶切图。操作如下:在刚才的窗口中点
restriction map 下面的 RMap setup 按钮,在出现的对话框中点 Add,再在出
现的对话框中点 select all,然 后 OK。点 sequence 下面的 sequence setup,
可以看到序列长度的设置等,在 color 栏中选 green,一路点 OK。此时显示如
下:
4
Page 5
生命经纬 www.biox.cn
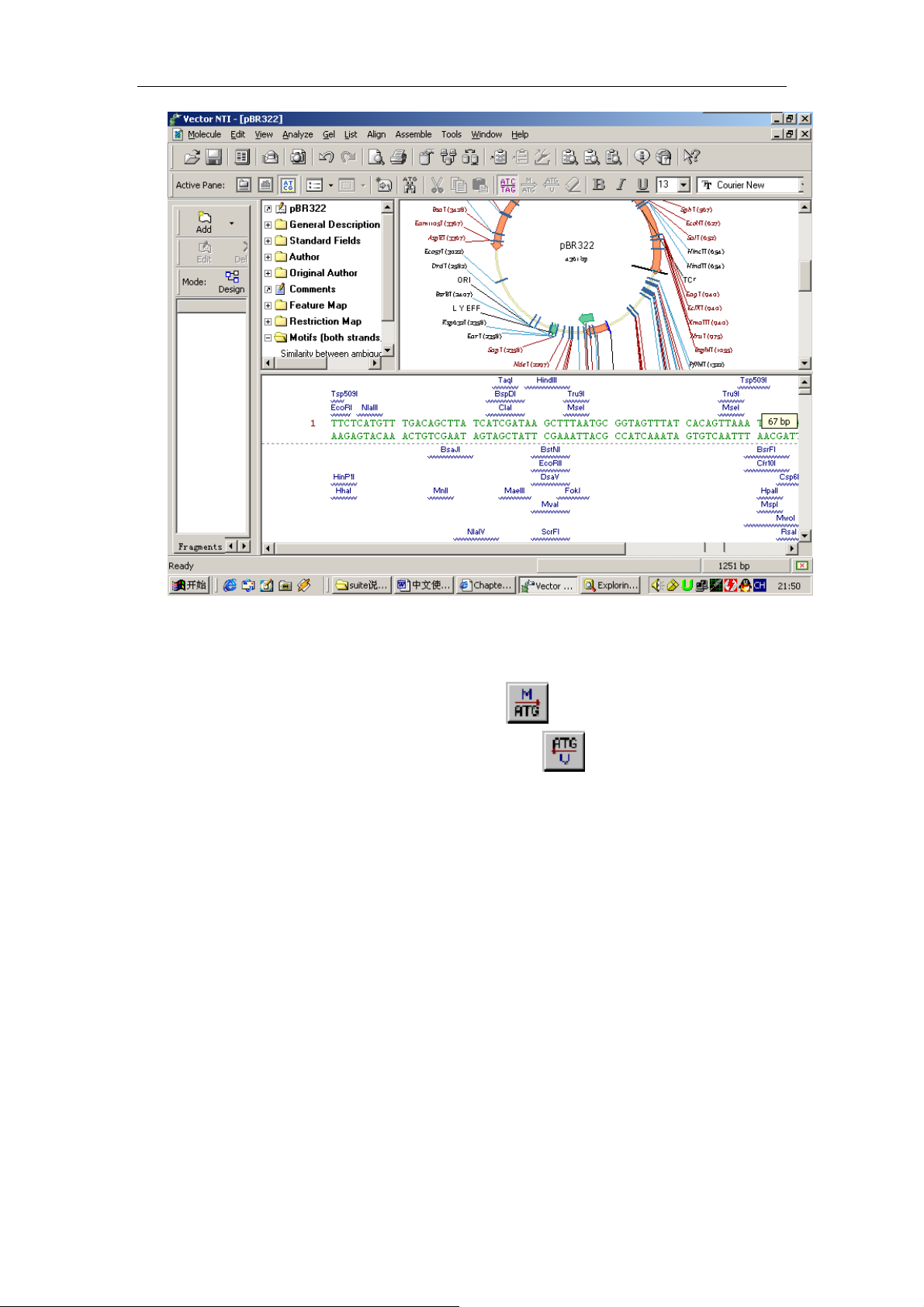
我们发现限制酶是大大的多了,不过美中不足的是序列显示的是两条链(正链和
互补链),实际上一条链就够了,还有最好再和编码的氨基酸一切显示。这好办:
先选中全序列(Ctrl-A),看到工具条中的
了没,点它。哈哈果然翻译成功了。不要忘了看
(Translate Direct)图标
左右两侧的图标哟,点点
看。(注意用户在发表文章的时候,一般在文章中发表自己克隆或表达的 DNA
序列,在 DNA 序列下面还有氨基酸序列,哈哈,这不帮你做到了。什么?内切
酶怎么去掉,刚才你怎么加上去的就怎么解除吧,看看我下面的图不就是办到
了):
5
Page 6
生命经纬 www.biox.cn
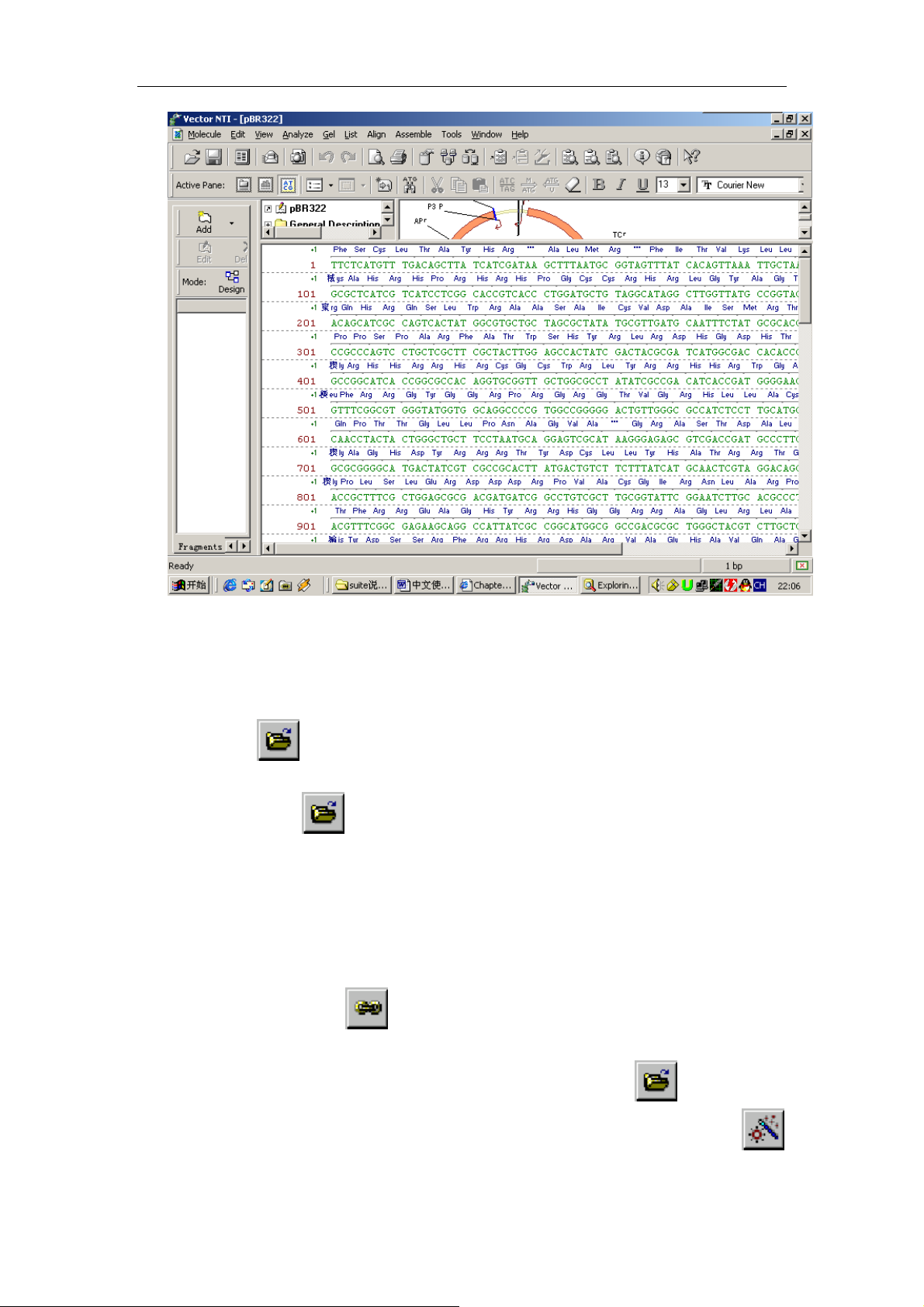
9、对 pBR322’s text 文本描述进行操作
拖动标尺,使文本区尽可能拉大。选中 Restriction Map 文件夹,然后找到
工具条中的
Expand Branch按钮,点它。其实这和双击 restriction map
文件夹是一样的,都是打开的意思,还有它左边的按钮。在找到 Feature Map
文件夹,然后按 按钮。看到 TC(R)了没,记住它。这是一个四环素抗性
基因,在第 7 章中将详细叙述如何将这段基因克隆到载体 PUC19 中。
10、将 pBR322’s 文本区和图区、序列区连接起来(可以让你一个一个的细
细品位 PBR322 的每个细小结构,全部看可能会眼花,那就一个一个的看吧)
激活文本区,然后找到
(Link Panes)按钮,点它。完了,PBR322的图
区上的任何标记都没了,成了一个圆圈。还有,序列区的酶切标记也没了。哈哈,
不用急,先点文本区的 Restriction Map 文件夹,然后点
按钮打开文件
夹里面的分支。现在看看,酶切标记又重新显示出来了。激活图形区,找到
(Standard Arrangement)按钮了没,点它。会发现酶切图谱显示的方式
6
Page 7
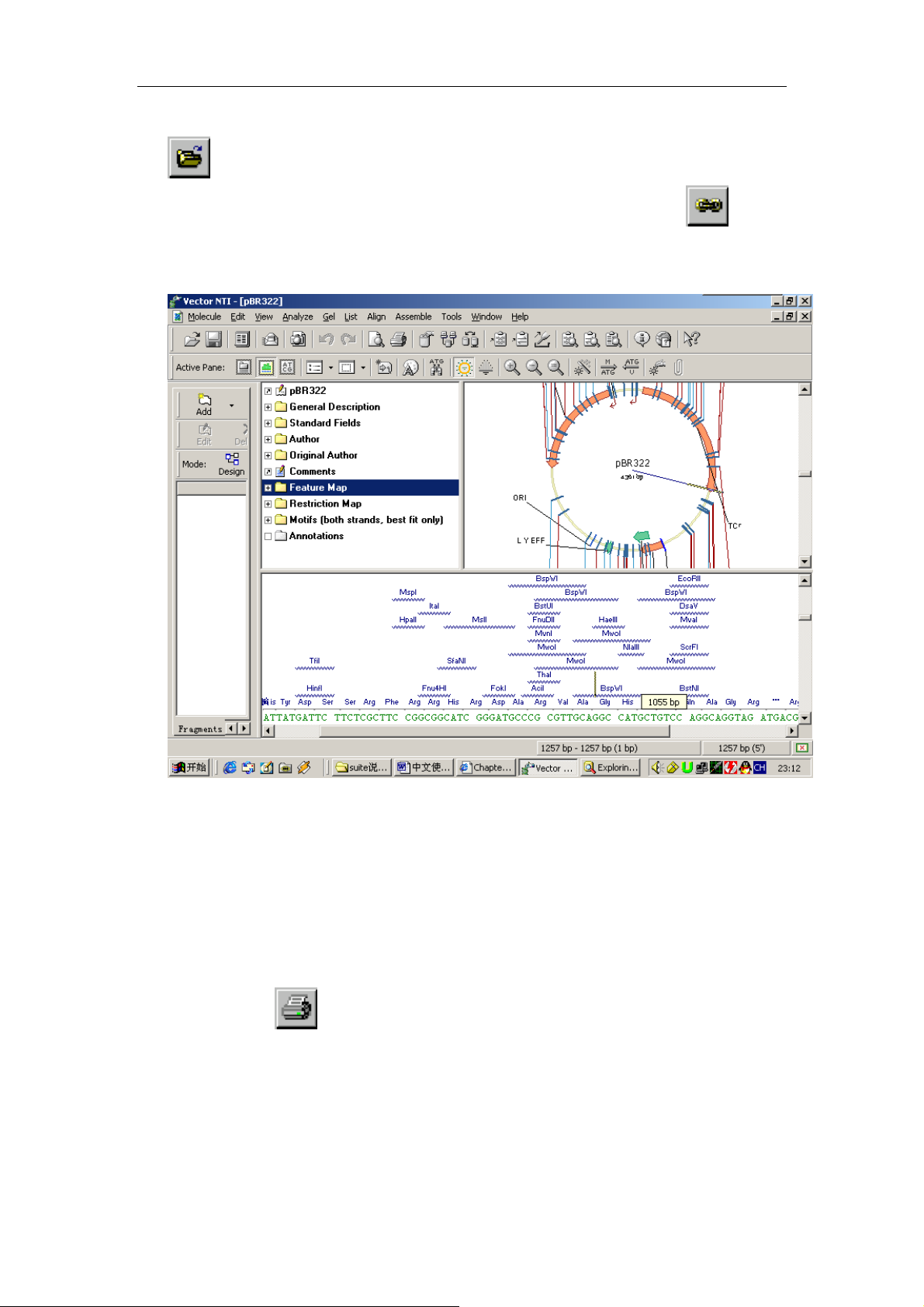
生命经纬 www.biox.cn
和刚才不一样了,这是标准方式。在文本区中选中 feature map 文件夹,点
打开,发现图形又变了。依次关掉 feature map 中的其他文件夹,只留
TCR,此时图中只有 TCR 一个标记了。最后别忘了再点一下链条
图又回到原样。如下图:
,发现
(看看上面的时钟都 23:12 了,该睡觉了),明天继续。
11、打 印 pBR322’s 文本 description, 图形 map, 和序列 sequence
打印文本:先激活文本区,(就是 active pane 右边的第一个按钮,或者直接
用鼠标在本文区点一下),然后按 expand branch 按钮打开文本区内的所有文
件夹,然后点
打印。同样要想打印图形或序列,先激活其所在的选区,然
后按打印机图标即可。
12、为 41BB_HUMAN 创建显示窗口
7
Page 8
生命经纬 www.biox.cn
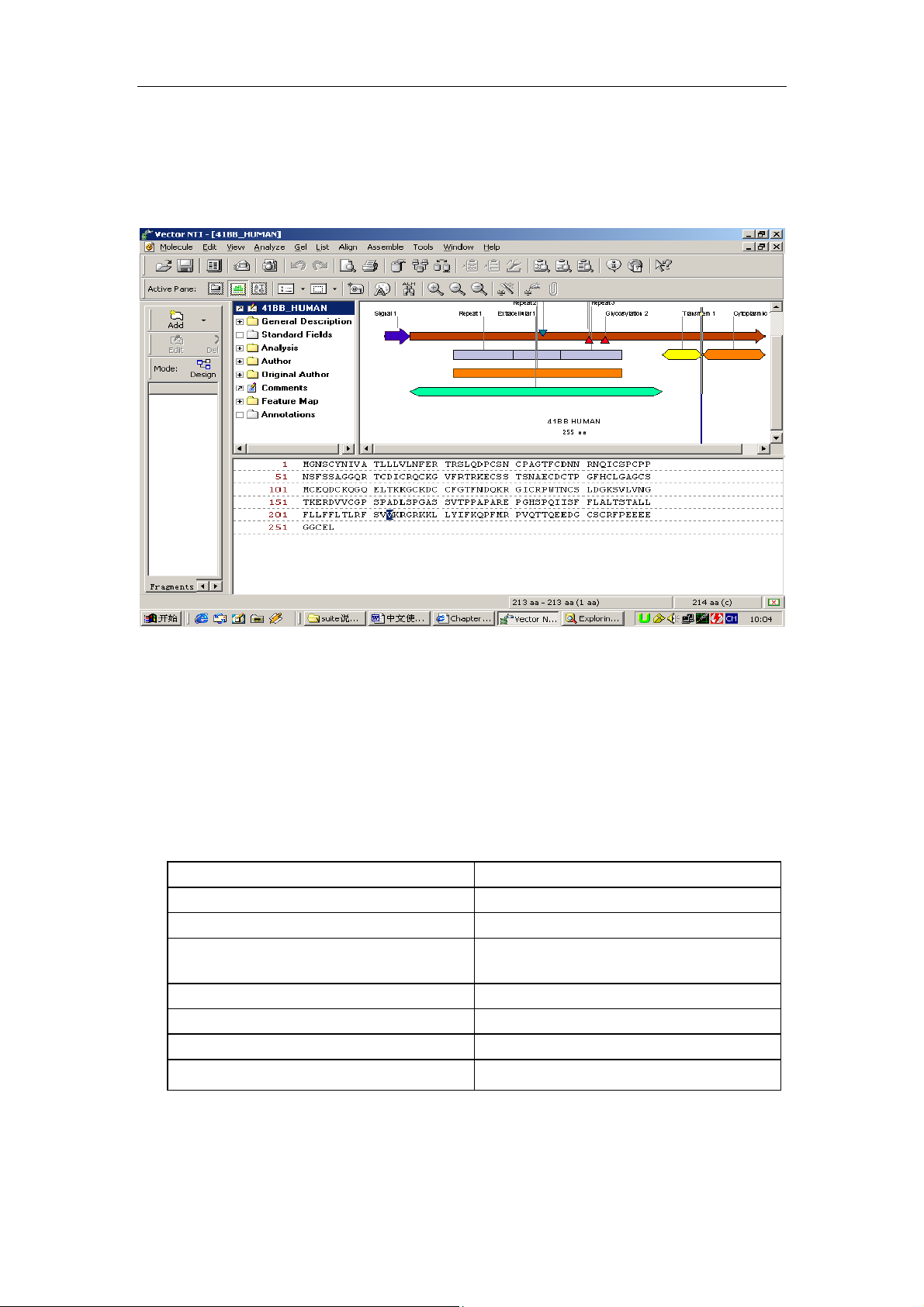
点击窗口下面的 exploring——local vector NTI database 图标,打开打开
Explorer 窗口,点击窗口左上角的下拉式菜单,选 Protein Molecules (MAIN)
数据库。找到 41BB_HUMAN’s 并双击。打开窗口如下:
窗口显示结构和 DNA 序列的显示窗口一致,也包括文本区,图形区和序列区三部
分。菜单和工具条也基本一样。在文本区中双击 Analysis 文件夹,则蛋白自动分析
结果以表格的形式在下面显示出来。下面我们把这两个表格拷贝到 word 文档中,
先用 shift+鼠标将两个表格选中,然后点工具条中的照相机(camera)命令,在
出现的对话框中可以看到序列的 range 中的 selection 已被选中,点 Copy.,然 后
打开一个 word 文档,粘帖(ctrl-V),则表格被完整的拷贝到 word 文档中了。如
下所示:
Length 255 aa
Molecular Weight 27897.66 m.w.
1 microgram = 35.845 pMoles
Molar Extinction coefficient 11250
1 A[280] corr. to 2.48 mg/ml
A[280] of 1 mg/ml 0.40 AU
Isoelectric Point 8.13
Charge at pH 7 3.72
8
Page 9

生命经纬 www.biox.cn
Amino Acid(s) Number count % by weight % by frequency
Charged
(RKHYCDE)
Acidic (DE) 25 10.85 9.80
Basic (KR) 29 14.43 11.37
Polar (NCQSTY) 90 34.08 35.29
Hydrophobic
(AILFWV)
A Ala 1 1 3.02 4.31
C Cys 25 9.33 9.80
D Asp 1 1 4.51 4.31
E Glu 14 6.34 5.49
F Phe 16 8.14 6.27
G Gly 21 4.85 8.24
H His 2 0.96 0.78
I Ile 7 2.83 2.75
K Lys 13 5.85 5.10
L Leu 21 8.48 8.24
M Met 3 1.38 1.18
N Asn 12 4.88 4.71
P Pro 18 6.38 7.06
Q Gln 12 5.40 4.71
R Arg 16 8.58 6.27
S Ser 22 7.12 8.63
T Thr 17 6.24 6.67
V Val 11 3.97 4.31
W Trp 1 0.63 0.39
Y T yr 2 1.12 0.78
B Asx 23 9.39 9.02
Z Glx 26 11.74 10.20
X Xxx 0 0.00 0.00
83 36.68 32.55
67 27.06 26.27
13、为 1B14_HUMAN 创建显示窗口
重新回到 exploring——local vector NTI database 窗口,找到
1B14_HUMAN 分子并双击打开。如下图:
9
Page 10

生命经纬 www.biox.cn
注意该蛋白分子图形上的各种特征显示的十分紧凑,大有眼花缭乱的感觉,为了
方便起见,我们可以按刚才介绍的 link 命令来逐一显示各个 feature。操作方
法和 DNA 分子一致。
关闭窗口,结束,当最后一个窗口关闭是屏幕提示 this will end your vector
NTI session,点确定(OK)关闭。
三、Chapter 2
Tutorial: Molecule Operations 分子操作
目的:对 pBR322 的 general data, feature map, and sequence 进
行编辑(注意蛋白质和 DNA 分子的操作是一样的)
1、登录 Vector NTI 程序(刚刚说完,不用再教了吧)
2、打开 pBR322 的显示窗口(再罗嗦一遍吧,程序 vector NTI——
Exploring local vector NTI Database—— DNA/RNA Molecules
(MAIN)——PBR322,双击。)
10
Page 11

生命经纬 www.biox.cn
3、对 pBR322’s 的常用数据(general data)进行编辑
在文本区的最上面,双击 PBR322 名字,弹出下面窗口:
st
1
:给PBR322 加关键词:点keywords,在弹出的关键词窗口中输入My own
plasmid,点Add。回到DNA/RNA Molecular 窗口,将最下面的
description中的内容替换成My pBR322。点OK(确定)。注意屏幕的左上
角pBR322*,在PBR322 的后面有一个星号,说明现在显示的是PBR322 的
修饰形式。现在我们要在数据库中保存这一结果:
菜单 molecular——save as,在弹出的对话框中输入序列的名字 My
pBR322,点 OK。这时发现星号不见了,说明结果已保存到数据库中,这时
数据库中关于 PBR322 的 DNA 分子有两个,一个是原始的 PBR322(就是
最初打开的那个),另一个就是我们保存的那个 my PBR322。
4、 编辑 My pBR322’s 序列
激活序列区,菜单 edit——set selection,在对话框中输入范围 21-40,点 OK。
会发现序列选区内含有 ClaI 和 HindIII 两个酶切位点。点击菜单 edit——
11
Page 12

生命经纬 www.biox.cn
new——Replace Sequence 21 bp–40 bp,将窗口中第 23 和 24 位的
TC 删除分别用 AA 代替,窗口将显示如下:
注意该窗口左下脚显示有:inserted 2,delete 2,什么意思都不用说了。点 OK,
注意序列中的 ClaI 位点立刻消失了。如下图所示:
5、将 My pBR322 的修改结果取消,不保存到数据库
12
Page 13

生命经纬 www.biox.cn
注意刚才修改完后,在屏幕左上角 My pBR322 的后面,有一个星号,说明当
前显示的分子已经修改,下面将修改结果取消,点击菜单 molecular——
Revert To Saved,点 OK 确定。此时数据库将刚才的修改结果取消了。(如
果需要保存修改结果则在 molecular 菜单中选 save as 命令)
6、如何插入新的序列片段
通常在编辑序列的时候需要在序列图谱中插入一段基因或者一段特征序列,先找
到序列中的 AP(R) 标志( 3293 bp–4156 bp)和 TC(R) 标志( 86
–1276 bp),菜单 edit——Set Caret Position,输入 200,将光标打到
200bp 处,下面我们要在此位置处输入 10 个 T 碱基。点菜单 edit——new—
—insert sequence 200bp,在出现的对话框中输入 10 个 T,点 OK,然后又
出现一个对话框,问你是否确认当前的序列已经修改(CDS TC(R) is
affected by sequence editing,Delete, Delete All, Keep, and
Keep All),点 keep.我们发现在 200bp 序列处多了 10 个 T。我们将鼠标移
到 AP(R)处,我们发现其位置已经顺时针移了 10 个碱基(3303 bp–4166
bp)。其实,在原始序列中一旦插入一段序列后,系统会自动改变图谱中各特
征的相对位置,如果插入的序列位于某一特征序列的内部,我们点 Keep,NTI
会自动向 3’端移动。现在我们将鼠标移到 TCR 处,发现其 3’端已经后移了 10
个碱基(1286),不过 5’端没动哟。如下图所示:
13
Page 14

生命经纬 www.biox.cn
7、编辑 TC(R) 信号特征
将鼠标移到图形的 TCr 处,当箭头变成“手”的形状时双击(或者点鼠标右键,
选 feature properties),在出现的对话框中用户可以修改 TCr 的位置、名称和
描述。我们将名称(name)由 TC(R)改成 Old TC(R),然后在最下面的
description 中输入描述:“10 bp fragment inserted”,点 OK。则
图形结构变成:
8、 删除 P2_P 信号,并加入新的序列特征
在图谱中找到 P2P,选中,点鼠标右键,选 Delete Feature From Fmap
(或者从 edit 菜单中选择此命令),此时 NTI 将提示 P2_P will be deleted
from the feature map,点 OK(确定)。我们发现图谱中 P2P 已经没有了。
下面我们我为 PBR322 序列加入一个新的特征:
edit——set selection——输入 3000-3500bp,点 OK。在工具条中找到
(add features)按钮,点它。(也可以从 edit——new——add feature
to FMap 进入)。在出现的对话框 feature name 中输入 New Feature,
注意 NTI 默认的特征类型(feature type)为 Misc. Feature,用户可以
14
Page 15

生命经纬 www.biox.cn
从列表中选择自己认可的特征。最后点 OK。如图:
下面将 My pBR322 的修改结果保存到数据库中:菜单 molecular——save
as,(如果不想改名的话)点 OK,NTI 将提示 My pBR322 已经存在,是否
覆盖,点 overwrite.
9、 修改 My pBR322 的起始坐标(这样可使刚才插入的 10bp 片段和最初 的 PBR322 具有一致的坐标系)
菜单 Molecule——operations——Advanced (DNA/RNA)—— Change
Starting Coordinate。在出现的对话框 new start(新的起始点位置)输入:11
(因为刚才插入了 10bp,所以起始点是 1+10=11)。点 OK,此时 NTI会提
示当前坐标已被修改,是否继续,点 OK 确定。现在我们会发现显示窗口中 TCR
的位置已经变成了(76bp–1276bp),还有 AP(R)的位置(3293 bp–4156
bp),这都和最初的 PBR322 一致。如下图:
15
Page 16

生命经纬 www.biox.cn
10、退出显示窗口,关闭 NTI
四、Chapter 3
Tutorial: Working With A Molecule’s Graphical Representation(对分子图形的
展示进行操作)
目的:以 DNA 分子为例,对分子图形的展示进行操作(蛋白质的操作和 DNA
类似),为 pBR322 图形创建一个展示窗口,并保存到分子文档中
1、登录 Vector NTI 程序
2、新窗口打开 PBR322(与前两章的打开方式略有不同)
在 NTI 主窗口中,点击工具条最左边的打开文件夹(或者 molecular——Open),
在弹出的 Open 窗口中点击 database DNA/RNAs,在列表中找到 PBR322,然
后 OK。
3、显示窗口的调整
比如我们想观察图象的详细情况,首先用标尺拖动图形窗口,至于序列和文本区,
可以很小,因为我们的目的是看图。然后点
满意为止,如果用户想一步步的看放大效果,可以按住 shift 的同时点
或 让图形放大或缩小,直到
。
4、修改图形的自动排列设置
按住 ctrl 的同时,点
(Standard Arrangement),用户可以根据弹出
的小菜单,来选择图形线条的粗细和字体的大小,直到满意为止。
5、修改图形的编码区信号( CDS signals)设置
点中图形中任意编码区(粗箭头),然后按鼠标右键,在弹出的下拉菜单中,选 CDS
Display Setup,用户可以在弹出的 Graphics Display Setup 对话框中修改
所选编码区的名称、颜色标记、箭头的粗细等,(实际上 NTI 默认的就很好了)。
用户也可以通过点 more 来进行更多的修改(比如字体和大小等,和 word 中字体
16
Page 17

生命经纬 www.biox.cn
的处理很相似),修改完后一路 OK 点下去即可。注意这种修改只是针对本窗口中
的分子,对其他分子没有影响。比如我们将箭头填充成兰色。下面我们保存这一设
置:点击
(display setup)右侧的小三角形符号,在弹出的菜单中选 Save
Settings As,然后在弹出的窗口中给刚才的设置风格取个名字,不妨叫 blue,
点 OK,注意此时 NTI 会提示是否保存其他没用过的风格,点 NO。现在再点击
(display setup)右侧的小三角形符号我们会发现 blue 已经在下拉菜单上了。
(注意这种改变一改就是好几个箭头一块改,如果只想改一个箭头的颜色,请看第
6 条)
6、打开图片编辑方式
NTI 提供了两种编辑图片的方式,分子编辑方式 (默认)和图片编辑方式。激活图
形区然后按
(edit picture)按钮。然后点中图形中任意标记或者箭头,按
住鼠标右键,在弹出的下拉菜单中选 properties(属性)或者 style(风格)等,进行
设置,注意此时设置的仅仅是所选的箭头或者标记,而不是整个分子(在第 5 条中
设的确是整个分子,请注意比较,也就是第 5 条的方法是分子编辑方式,两种方式
的转换通过
按钮)。
7、将 TC(R)箭头变成兰色网格
操作如下:点中
按钮,点中 TCR 箭头,按住鼠标右键,选 properties——
fill,按右图方式选择:
点 OK(如果是中文操作系统,OK=确定)
17
Page 18

8、放大 TC(R)箭头
生命经纬 www.biox.cn
点中 TCR 后,当鼠标在箭头附近移动时,会出现两种十字形标记:
过鼠标拖动
可以改变箭头的胖瘦,而拖动 则可以改变箭头的相对位置(移到
和 ,通
圆内或圆外)。如果想让箭头按圆圈转动,可以在按住 ctrl+shift 同时,拖动
注意这种拖动并不能修改分子本身,仅仅是改变位置而已,如果想修改分子中的标
记,请使用第二章介绍的方法。现在,拖来拖去是不是将箭头拖的乱七八糟了,那
就按
取消吧。如下图:
。
9、修改 TC(R)标记的格式
将鼠标移到 TCr 标记上,双击。显示下面的对话框(就是属性)
18
Page 19
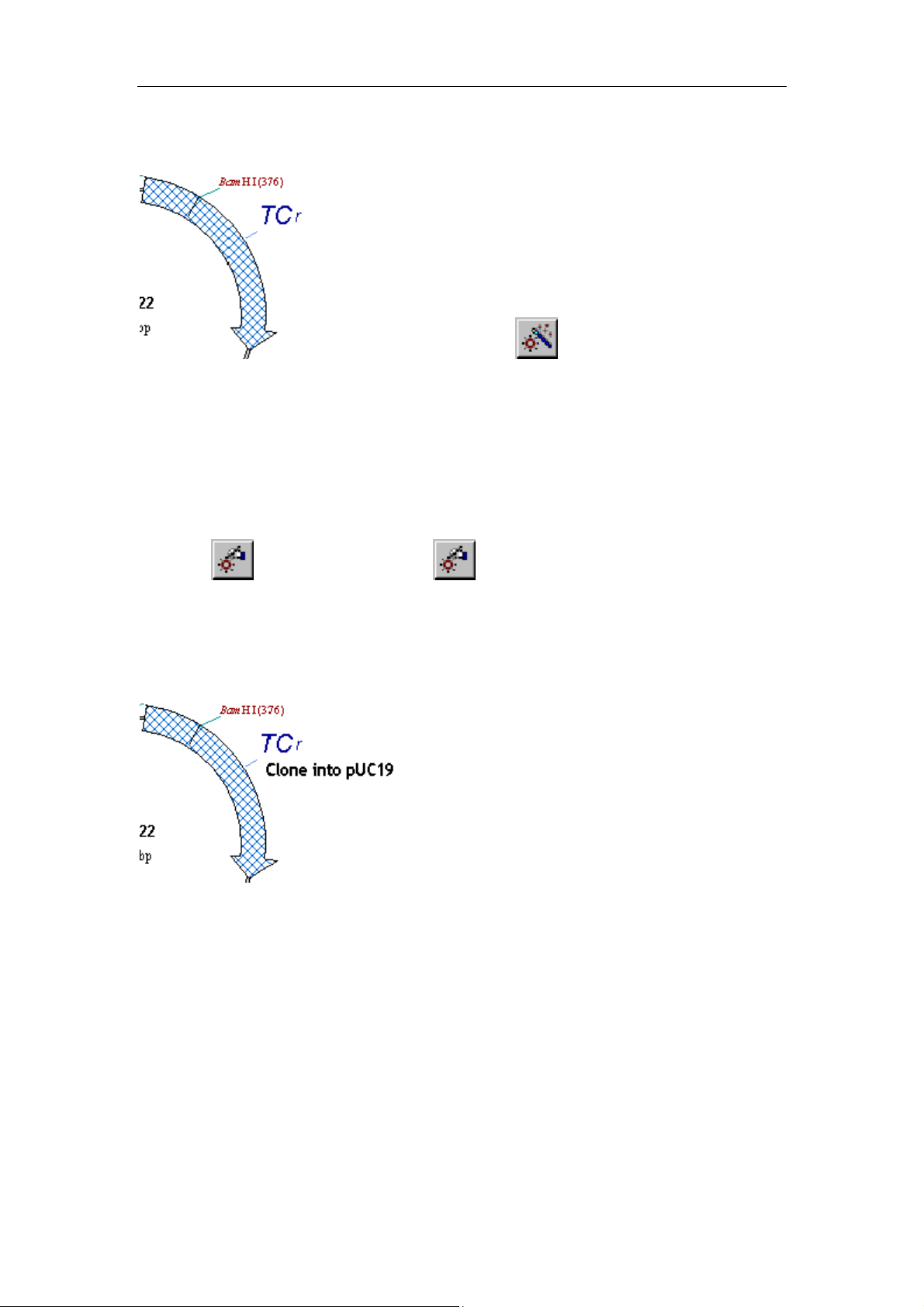
生命经纬 www.biox.cn
点 Font,选择斜体 18 号字,点 OK。TCR 将显示如下图:
我们发现,字体大了。点 (Standard
Arrangement)按钮,使标记的信号回到标准位置,如果图片已经修改,NTI
还会不厌其烦的提示是否继续,点确定吧。
10、加入文本注释
看到窗口工具条最右侧的“回形针”符号了没,点它(说什么,没反应?哈哈,那
你肯定把
忘了,如果没反应,点 后再点回形针肯定行),在弹出的对话
框中输入“Clone into pUC19”,点 OK。此时加入的注释出现在圆圈的中央,
用鼠标拖到 TCr 的下面即可(什么?字体太小,哈哈,点中鼠标右键从 properties
中选择字体的大小和颜色)。如下图:
如果觉得不爽,可以先选中刚才添加的文本,点菜单 edit——Delete Annotation
进行删除。注意以上修改的仅仅是显示的界面,NTI 并不能将修改的显示结果保存
到数据库中,所以用户也可以发现在左上角并没有*号标记,也就是数据库中的分
子没有改变,如果想让它改变的话,只能将文件存到文档文件中了。
11、将 pBR322 分子显示结果保存到文档文件中
菜单 Molecule——save as——save as file,名称 NTI 已经给起好了,就是
pBR322.gb。选择好要保存的目录,点 OK。如果感觉刚才创建的 blue 比较爽,
19
Page 20

生命经纬 www.biox.cn
可以在保存前选择 右侧的三角按钮选择 blue。此时系统会提示是否创建
html 文件描述分子,点 yes(这样我们可以和朋友通过 internet 进行交流了)。
注意该文档完全与数据库相关联,其显示的信息与数据库中的信息一致。但风格与
数据库不同,比如箭头的颜色、字体的大小等(当然由用户决定)。现在用户可以
先关闭文档然后再打开就可以发现显示的风格和数据库中的风格完全不同(不过内
容还是一样的)。注意:对于原先数据库中没有的分子(比如用户根据自己的需要
自己构建或从 GENBANK 下载的,并不能直接修改显示风格,如果想修改其显示
风格,先将该分子保存到数据库中,这样就可以了)
退出程序
五、Chapter 4
Vector NTI 工具和 internet 连接
目的:将本地数据发送到 internet 上,并进行 BLAST 搜索, 序列对排、比较和分析等
1、登录 Vector NTI
2、在新窗口 打开 pBR322
3、通 过 exploring local vector NTI database 窗口(切记)选中 pBR322
整个分子并利用 BLAST 搜索工具进行序列比较(注意:如果不是通过 exploring
窗口,而是通过 NTI 主窗口“打开”命令,打开 PBR322,则最终的搜索结果不
会直接显示在屏幕上,而是发到用户指定的 email 中,但结果都是一样的)
选中后,点 exploring local vector NTI database 窗口中的 Tools——
Compare Against——GenBank via BLAST on NCBI Server ,在弹出的
菜单中按下面窗口选择全序列和正链并点 OK 确认(注意保证网络连接正常,否则
无法连接到 Genbank):
20
Page 21

生命经纬 www.biox.cn
当连接成功后,将弹出 NCBI 入口口,如下图所示(不过由于使用的 internet 浏览
器不同,可能每个人显示的窗口可能不一样,我使用的示 IE5.0):
点 blast,弹出如下窗口:
点 format,此时 NCBI 将正式开始搜索(注意这个过程可能需要几分钟,请耐心
等待,窗口下面会显示该过程需要的时间,比如 This page will be
21
Page 22

生命经纬 www.biox.cn
automatically updated in 105 seconds until search is done),结果
显示如下:
用户可以通过拖动滚动条查看结果(很多的),找到gi|416343|gb|U03501.1|YRP7 Yeast
replicating vector YRp7, c...
(Genbank格式),如下图所示:
8528 0.0 了没,点它。屏幕将显示此克隆载体的详细信息
22
Page 23
生命经纬 www.biox.cn
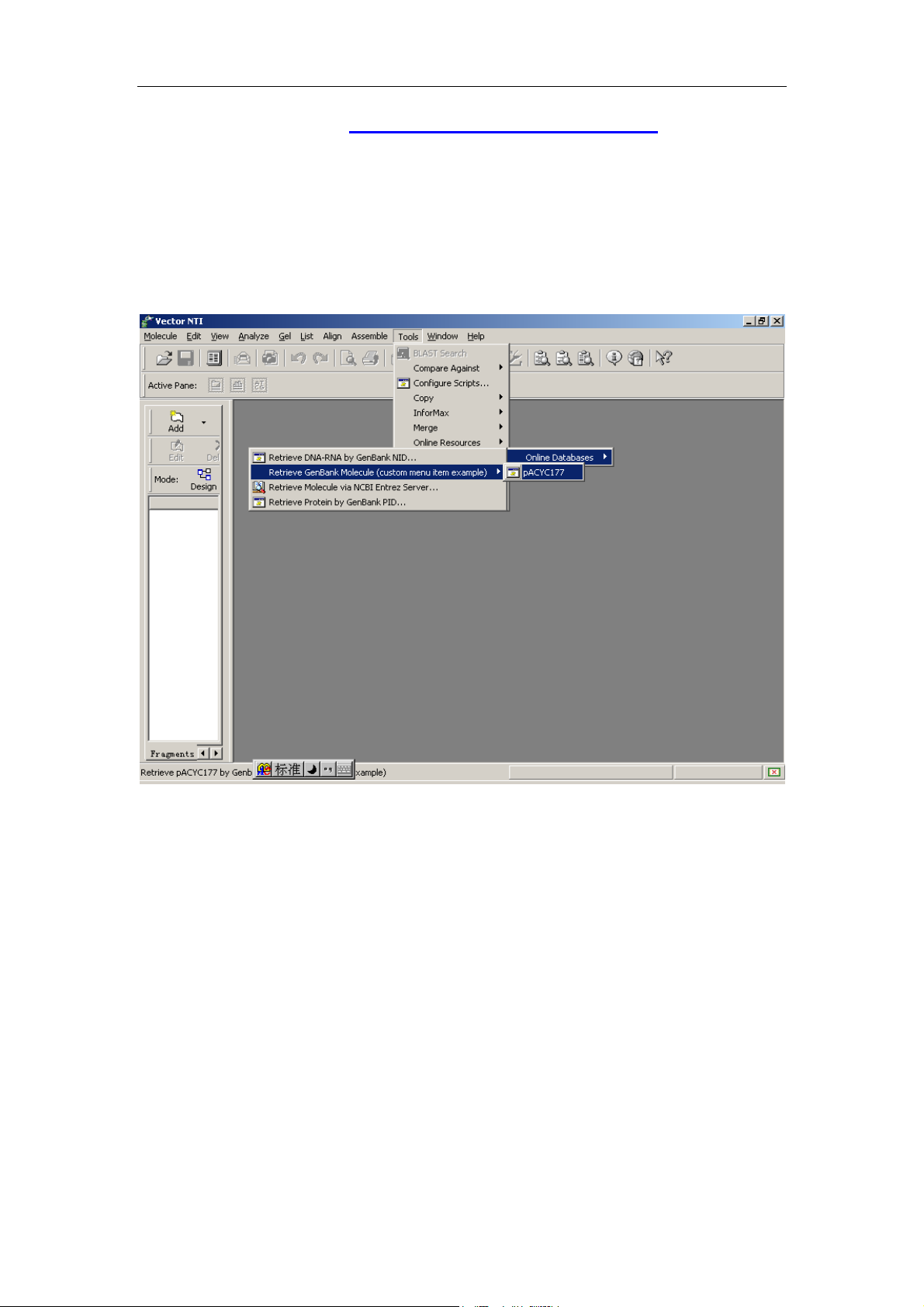
4、 刚才的搜索结果gi|416343|gb|U03501.1|YRP7转化成NTI文档
格式:操作如下:
在浏览器中点:编辑——全选,然后编辑——复制,将屏幕拷贝到键盘上(或者直
接点击窗口中的 add to clipboard)。回到 NTI 主窗口,点:tools——open
——GenBank From Clipboard——online database,按下图所示:
选中 Pacyc177 后,Genbank 文本自动以 NTI 格式显示出来(注意,如果用
户安装了 simvector 软件后,那就用不着 NTI 费心了,sim vector 会帮我们
自动打开),如下图(是 simvector 帮俺打开的,和 NTI 格式一样,不过感觉
更爽):
23
Page 24

5、利用序列比较工具 alignment
生命经纬 www.biox.cn
在 exploring local vector NTI database 窗口中,选择 protein
molecular 数据库,利用 shift+鼠标选中 41BB_HUMAN 和
41BB_MOUSE,然后点击菜单 align——Multiple Sequences on BCM
Server ,NTI 将以 FASTA 格式打开搜索窗口,点 submit,如果网络连接正
常,多序列比较结果显示如下:
24
Page 25

生命经纬 www.biox.cn
下面我们利用 ExPASy 服务器上的 ProtScale 程序分析结果,先回到
exploring local vector NTI database 窗口中,选中 41_HUMAN 分子,然
后点击菜单 Analyze——Protein Scales (hydrophobicity etc.) via
ProtScale on ExPASy Server,NTI 将打开蛋白质分析窗口,点 submit,
如果网络连接正确,则结果显示在如下窗口中:(用户可以通过此窗口查看该蛋白
所有二级结果信息,很爽的,还有很酷的图形呢)
6、保存搜索结果
25
 Loading...
Loading...