Page 1

Vector NTI User Manual
Vector NTI User's Manual
前言(INTRODUCTION) ....................................................................3
第一章 DISPLAY WINDOWS..............................................................4
一. 顯示 DNA 序列及圖形...........................................................5
二. 顯示蛋白質序列的方式.......................................................10
第二章 MOLECULE OPERATIONS................................................13
一. 對 PBR322’S 的常用資料(GENERAL DATA)進行編輯....13
三. 插入新的序列片段..................................................................15
四. 編輯 TC(R) 圖示 .................................................................16
五. 刪除 P2_P 標示,並加入新的序列特徵標示....................17
六. 修改 MY PBR322 的起始座標.............................................18
第三章 GRAPHICAL REPRESENTATION ....................................19
一. 爲 PBR322 圖形建立一個展示視窗....................................19
二. 修改圖形的自動排列設置...................................................19
三. 修改圖形的編碼區信號( CDS SIGNALS)設置 ...............19
四. 打開圖片編輯方式 ...............................................................20
五. 修改 TC(R)圖形....................................................................20
六. 加入註解注釋 .......................................................................22
七. 將 PBR322 分子顯示結果保存到文檔文件中...................22
第四章 BLAST SEARCH AND BLAST VIEWER ..........................24
一. 簡介 .......................................................................................24
二. BLAST 搜尋視窗..................................................................24
三. BLAST SEARCH RESULTS .......................................................28
四. SAV I N G BLAST SEARCH RESULTS ..........................................29
- 1 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 2

Vector NTI User Manual
第五章 ALIGNX ..................................................................................31
一. 開啟與執行: .......................................................................31
二. 參數設定與調整: ...............................................................35
三. 資料輸出與列印 ...................................................................39
第六章 ALIGNX-BLOCKS ................................................................40
一. 開啟與執行: .......................................................................40
二. 參數設定與調整: ...............................................................42
三. 資料輸出與列印......................................................................44
第七章 BIOPLOT ................................................................................45
一. 開啟與執行: .......................................................................45
二. 資料輸出與列印 ...................................................................49
第八章 CONTIGEXPRESS ................................................................50
一. INTRODUCTION ........................................................................50
二. PROJECT EXPLORER .................................................................50
三. WORKING IN FRAGMENT WINDOW ..........................................54
四. WORKING IN THE CONTIG WINDOW.........................................60
第九章 MISCELLANEOUS VECTOR NTI TOOLS ......................63
一. INTRODUCTION ........................................................................63
二. PUBMED/ENTREZ SEARCH.......................................................63
三. CITATION VIEWER ....................................................................66
附記:如何執行反安裝……………………………………………….69
- 2 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 3

Vector NTI User Manual
前言(Introduction)
程式附帶的資料庫(Vector NTI database)包括:DNA/RNA 序列、
蛋白質序列、限制酶、寡核苷酸、電泳 marker。此外程式還提供資料
庫開發(Database Explorer)功能,使用者可以自己修改、添加、拷
貝感興趣的各類資料庫。
建立新序列資料(有四種方法)
用 GenBank/GenPept, EMBL/SWISS-PROT 或 FAS TA、ASCII
等格式輸入 DNA 或氨基酸序列。
以 Copy/Paste 方式貼入,然後存到資料庫中。
從其他序列檔、linker、載體中剪切、拼接而成新序列
從 DNA 或 RNA 序列轉譯成蛋白質序列
關於新序列的內部特徵圖譜(例如 CDS、motif 等資訊),若是利
用 GenBank/GenPept, EMBL/SWISS-PROT or FASTA 等格式輸入的
序列,因為內含註解,所以都能直接顯示,但自己手工粘帖的沒有,
需要自己編輯
- 3 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 4
Vector NTI User Manual
第一章 Display Windows
目的:建立顯示視窗,並對載體圖、序列及註解進行操作
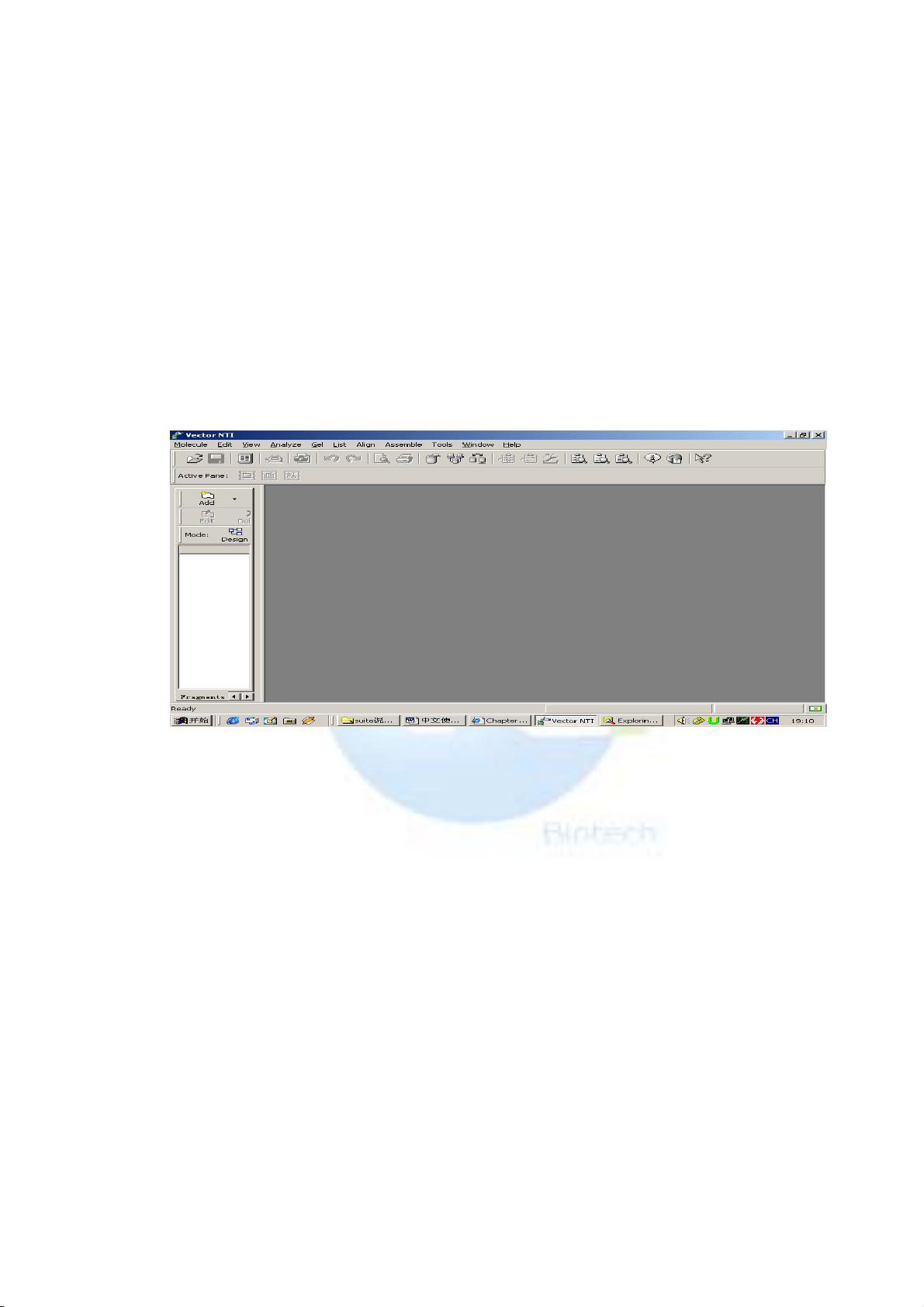
1.登錄 Vector NTI
安裝後首次登錄,系統將提示是否允許將新資料填入 Vector NTI
資料庫中,點 OK。這 樣 DNA molecules, proteins, enzymes, oligos,
and gel markers 將組成 NTI 的資料庫。並出現下列兩個窗口。
2. 觀察出現的 Vector NTI 工作視窗 和 Database Explorer 視窗
這個視窗爲工作視窗,由功能表欄和工具列兩欄,移動滑鼠到工
具欄任意選項處,滑鼠將會自動顯示每個工具列的功能。
- 4 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 5
Vector NTI User Manual
這個視窗爲 Database Explorer 視窗——local vector NTI database,
顯示的是上次打開的 DNA/RNA 或蛋白分子。
一. 顯示 DNA 序列及圖形
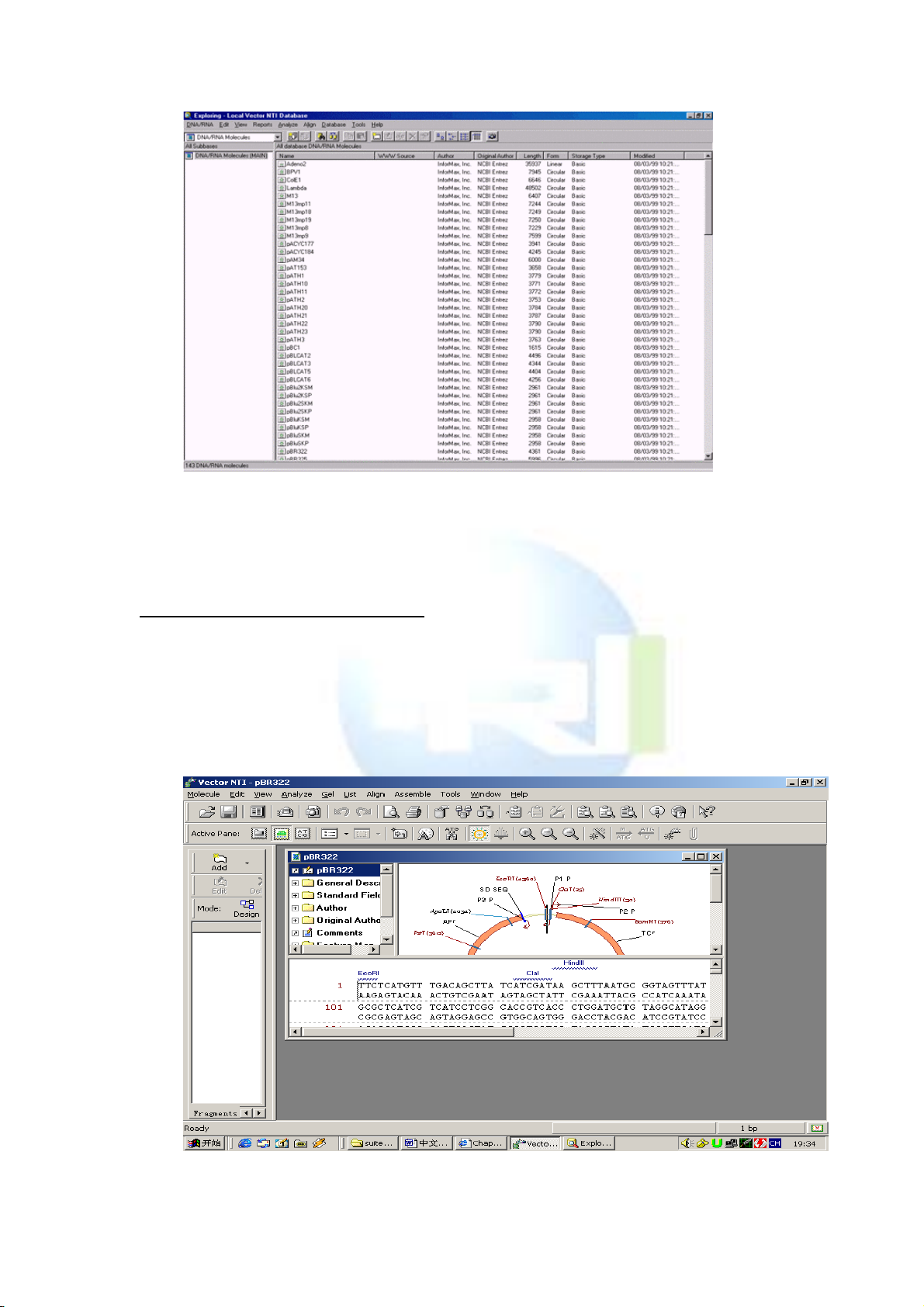
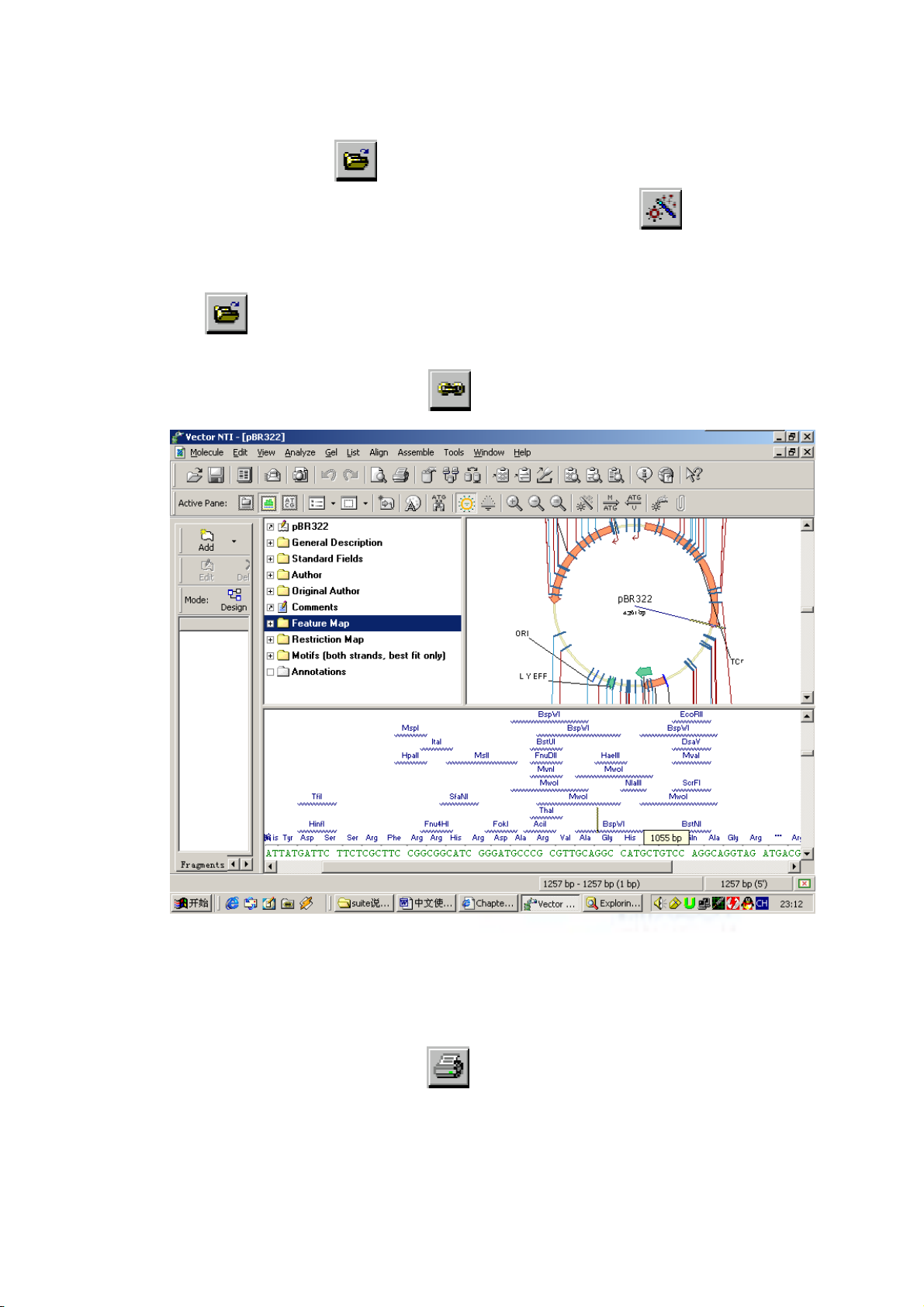
1. Create a Display Window for pBR322
打開 Database Explorer 視窗——local vector NTI database 視窗中
的 DNA/RNA Molecules (MAIN) 資料庫,找到 pBR322 分子並雙擊打
開。顯示如下視窗:
- 5 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 6

Vector NTI User Manual
2. . 觀察 pBR322 顯示視窗
上面的視窗由註解區(對該分子資訊的文字描述,雙擊文件夾可
以看到)、圖形區(標住限制酶位置等)和序列區(序列本文及限制
限制酶切位位)三個部分組成。
3. 顯示視窗的管理(透過拖拉尺規,改變視窗、或每個顯示區
的相對大小)
4. 轉換 pBR322’s 圖形區:在工具欄左邊的 active pane 右側有
三個按鈕,用滑鼠點當中那個(graphics pane)
5. 對 pBR322’s 結構圖進行操作:使用者可以嘗試點選 、
、 ,此時圖形的大小會發生變化。選取設定功能表
Editset selection,輸入 100bp–1000bp,然後按 OK.可以看到
所輸入的選取區間在圖形中以扇型框圈了起來.將滑鼠移到選
取的 5’端,可以看到
,通過拖拉可以延長或縮短選區範
圍,同樣在 3’端也能做到。如果使用者一次只想移動一個鹼
基用直接拖拉就可能不方便,假如使用者想在 5’端移動一個
鹼基,首先將滑鼠放到
處,然後按住 shift 和鍵盤右側的
或箭頭,則可以按一次箭頭移動一個鹼基距離。如果一
次想移動 10 個鹼基,則同時按住 shift+ctrl+箭頭。如果使用
者只是粗略選擇,可以直接將滑鼠移到圖中,用十字花拖動。
將滑鼠移到圖的 TCr 處,此時滑鼠箭頭變成
,並顯示 TCr
代表的含義,如果使用者此時按下滑鼠,則 TCr 的 coding
region 將被選取。
6. 檢查 pBR322’s nucleotide 序列
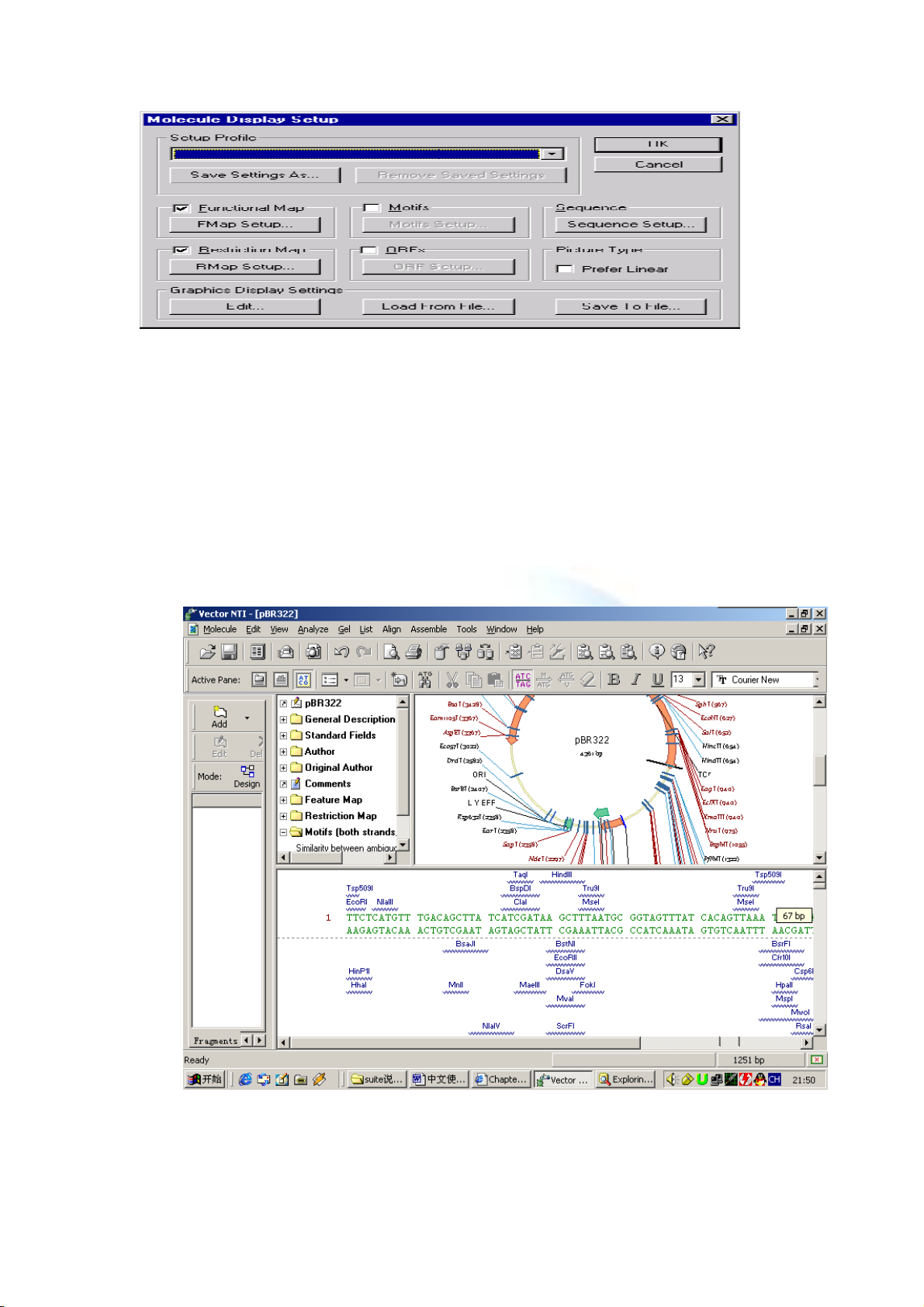
移動尺規,盡可能將更多的 PBR322 序列顯示出來,並點選序列
中任何一處。現在對序列顯示的樣式進行設定:點選
(Display
Setup )按鈕,顯示 molecular display setup 對話方塊:
- 6 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 7
Vector NTI User Manual
使用者可以對序列的顔色、大小、10 個一組顯示還是 15 個一組
(原始設定是 10 個鹼基一組),結構圖的顔色、限制酶圖譜(注意剛
開始顯示的 PBR322 上限制酶並不多)、ORF、Motif 等進行設置。現
在我們打算把序列字體的顔色由黑色變成綠色,顯示全部 PBR322 的
限制限制酶切位位。操作如下:在剛才的視窗中點 restriction map 下
面的 RMap setup 按鈕,在出現的對話方塊中點 Add,再在出現的對
話方塊中點 select all,然後 OK。點 sequence 下面的 sequence setup,
可以看到序列長度的設置等,在 color 欄中選 green,一路點 OK。此
時顯示如下:
可以發現所顯示的限制限制酶切位位變多了,不過美中不足的是
- 7 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 8
Vector NTI User Manual
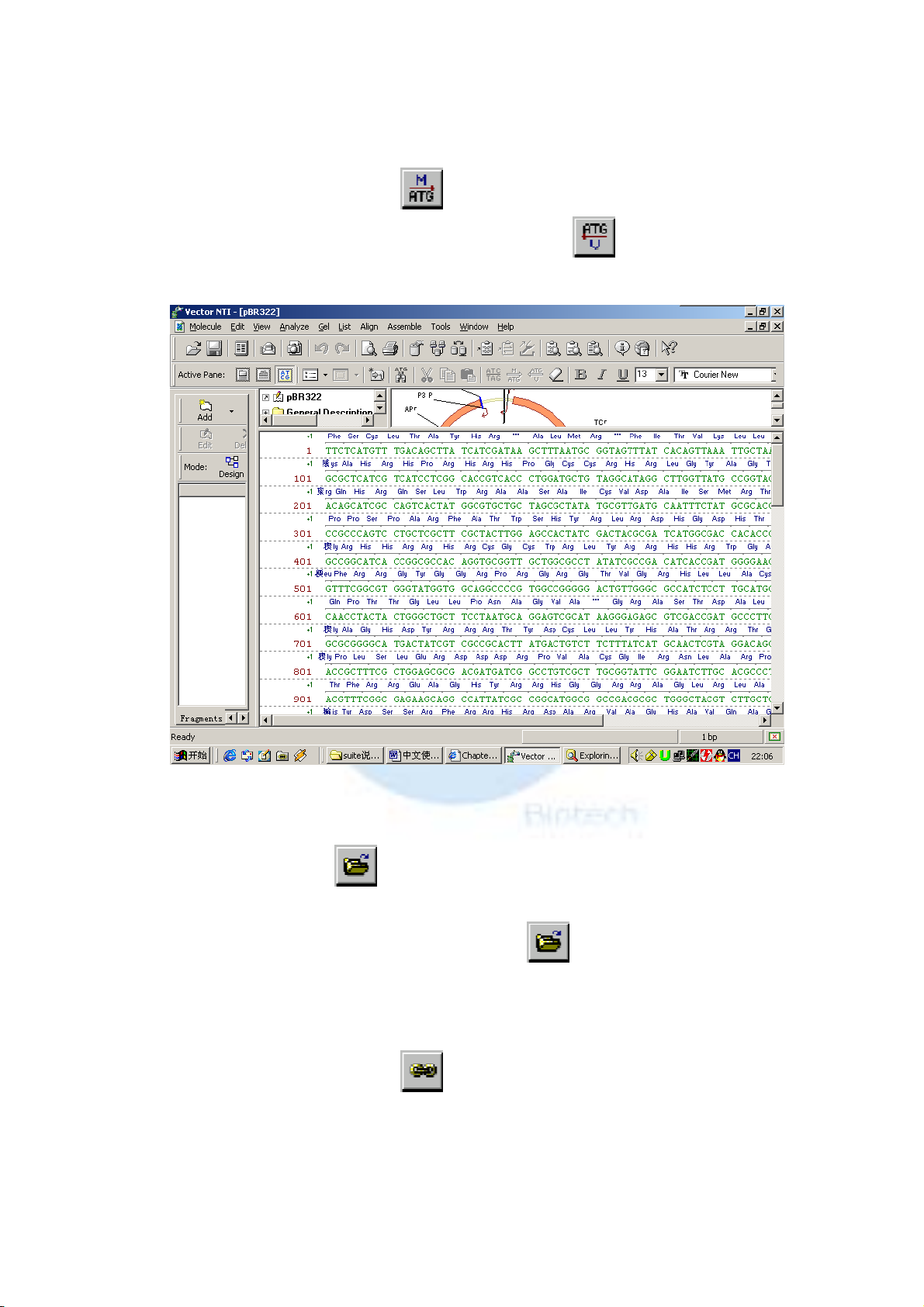
序列顯示的是兩條股(正股和互補股),實際上一條股就夠了,還有
最好再和編碼的氨基酸一起顯示。修改的方式如下:先選取全序列
(Ctrl-A),點選工具列中的
序列轉譯成蛋白質序列。此外也可以試著點選
(Translate Direct)圖示即可將 DNA
左右兩側的圖示。
若是覺得限制酶的切位會干擾顯示的情形,也可以將它去掉,如下圖
7. 對 pBR322’s text 註解描述進行操作
拖動尺規,使註解區盡可能拉大。選取 Restriction Map 文件夾,
然後找到工具列中的
Expand Branch 按鈕,點它。其實這和雙擊
restriction map 文件夾是一樣的,都是打開的意思,還有它左邊的按
鈕。在找到 Feature Map 文件夾,然後按
按鈕。可以看到 TC(R),
這是一個四環素抗性基因。
8. 將 pBR322’s 註解區和圖區、序列區連接起來
啟動註解區,然後找到
(Link Panes)按鈕,點選它可以將
PBR322 的圖區上的所有標記都去除,成了一個圓圈。還有,序列區
- 8 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 9
Vector NTI User Manual
的限制酶切位標記也沒了。要重新顯示則請先點註解區的 Restriction
Map 文件夾,然後點
制酶切位又重新顯示出來了。啟動圖形區,找到
按鈕打開文件夾裏面的分支。現在看看,限
(Standard
Arrangement)按鈕了沒,點它。會發現限制酶切位圖譜顯示的方式
和剛才不一樣了,這是標準方式。在註解區中選取 feature map 文件
夾,點
打開,可以發現圖形又變了。再依次關掉 feature map 中
的其他文件夾,只留 TCR,此時圖中只有 TCR 一個標記了。若是希
望圖形回到原樣,可以點選鏈條
,如下圖:
9. 列印 pBR322’s 註解 description ,圖形 map , 和序列
sequence
列印註解:先啟動註解區,(就是 active pane 右邊的第一個按鈕,
或者直接用滑鼠在註解區點一下),然後按 expand branch 按鈕打開註
解區內的所有文件夾,然後點
列印。同樣的方式,若要想列印圖
形或序列,先點選其所在的選取區,然後按印表機圖示即可。
- 9 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 10
Vector NTI User Manual
二. 顯示蛋白質序列的方式
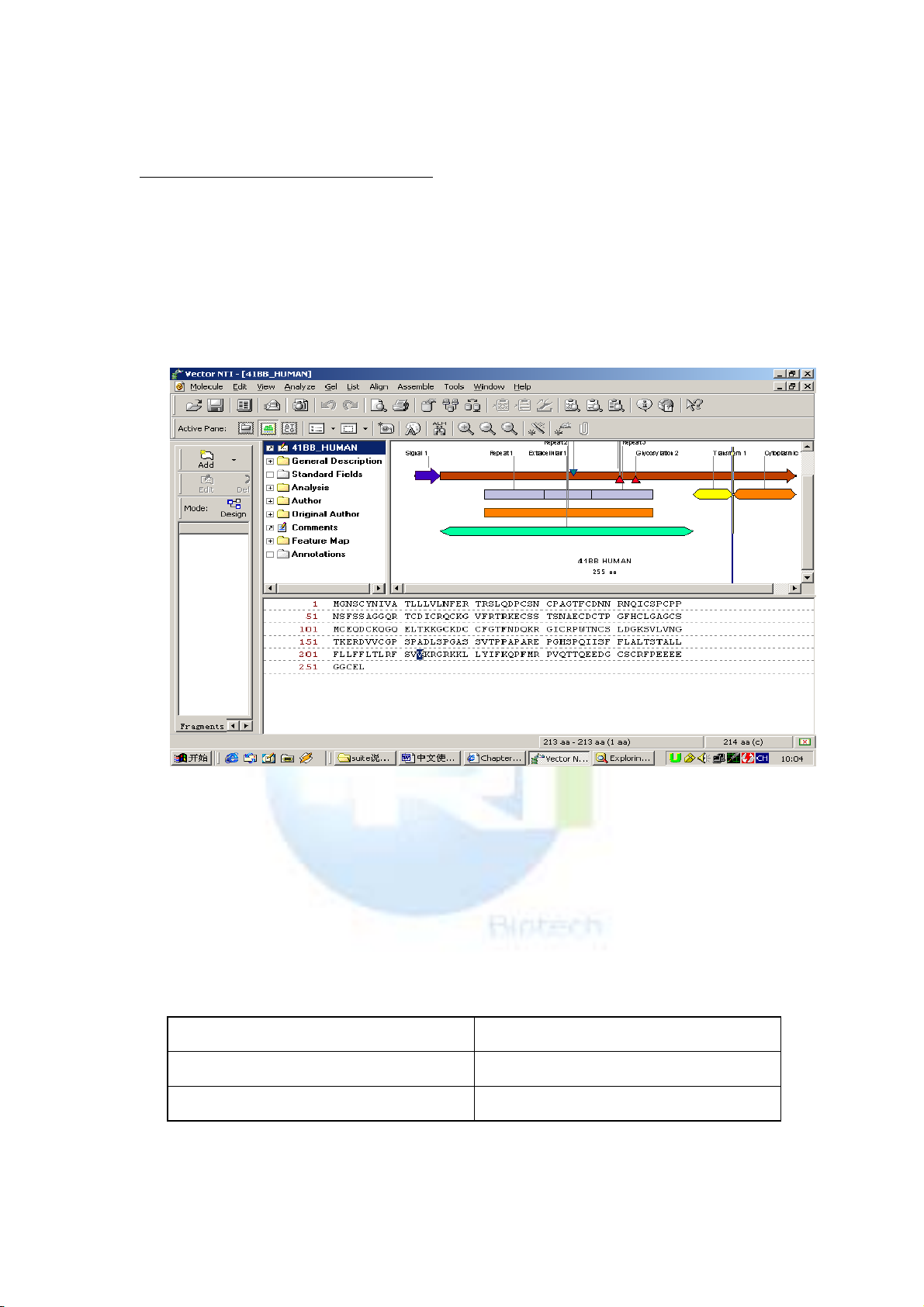
1. 開啟 41BB_HUMAN
點選視窗下面的 Database exploring——local vector NTI database
圖 示,打 開 Explorer 視窗,點選視窗左上角的下拉式功能表,選 Protein
Molecules (MAIN)資料庫。找到 41BB_HUMAN’s 並雙擊。打開窗口
如下:
視窗顯示結構和 DNA 序列的顯示視窗一致,也包括註解區,圖
形區和序列區三部分。功能表和工具列也基本一樣。在註解區中雙擊
Analysis 文件夾,則蛋白自動分析結果以表格的形式在下面顯示出來。
下面我們把這兩個表格拷貝到 word 文檔中,先用 shift+滑鼠將兩
個表格選取,然後點工具列中的照相機(camera),在出現的對話方
塊中可以看到序列的 range 中的 selection 已被選取,點 Copy.,然後
打開一個 word 文檔,按貼上(ctrl-V),則表格被完整的拷貝到 word
文檔中了。如下所示:
Length 255 aa
Molecular Weight 27897.66 m.w.
1 microgram = 35.845 pMoles
- 10 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 11

Vector NTI User Manual
Molar Extinction coefficient 11250
1 A[280] corr. to 2.48 mg/ml
A[280] of 1 mg/ml 0.40 AU
Isoelectric Point 8.13
Charge at pH 7 3.72
Amino Acid(s) Number count % by weight % by frequency
Charged
83 36.68 32.55
(RKHYCDE)
Acidic (DE) 25 10.85 9.80
Basic (KR) 29 14.43 11.37
Polar (NCQSTY) 90 34.08 35.29
Hydrophobic
67 27.06 26.27
(AILFWV)
A Ala 11 3.02 4.31
C Cys 25 9.33 9.80
D Asp 11 4.51 4.31
E Glu 14 6.34 5.49
F Phe 16 8.14 6.27
G Gly 21 4.85 8.24
H His 2 0.96 0.78
I Ile 7 2.83 2.75
K Lys 13 5.85 5.10
L Leu 21 8.48 8.24
M Met 3 1.38 1.18
N Asn 12 4.88 4.71
P Pro 18 6.38 7.06
Q Gln 12 5.40 4.71
R Arg 16 8.58 6.27
S Ser 22 7.12 8.63
T Thr 17 6.24 6.67
- 11 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 12
Vector NTI User Manual
V Val 11 3.97 4.31
W Trp 1 0.63 0.39
Y Tyr 2 1.12 0.78
B Asx 23 9.39 9.02
Z Glx 26 11.74 10.20
X Xxx 0 0.00 0.00
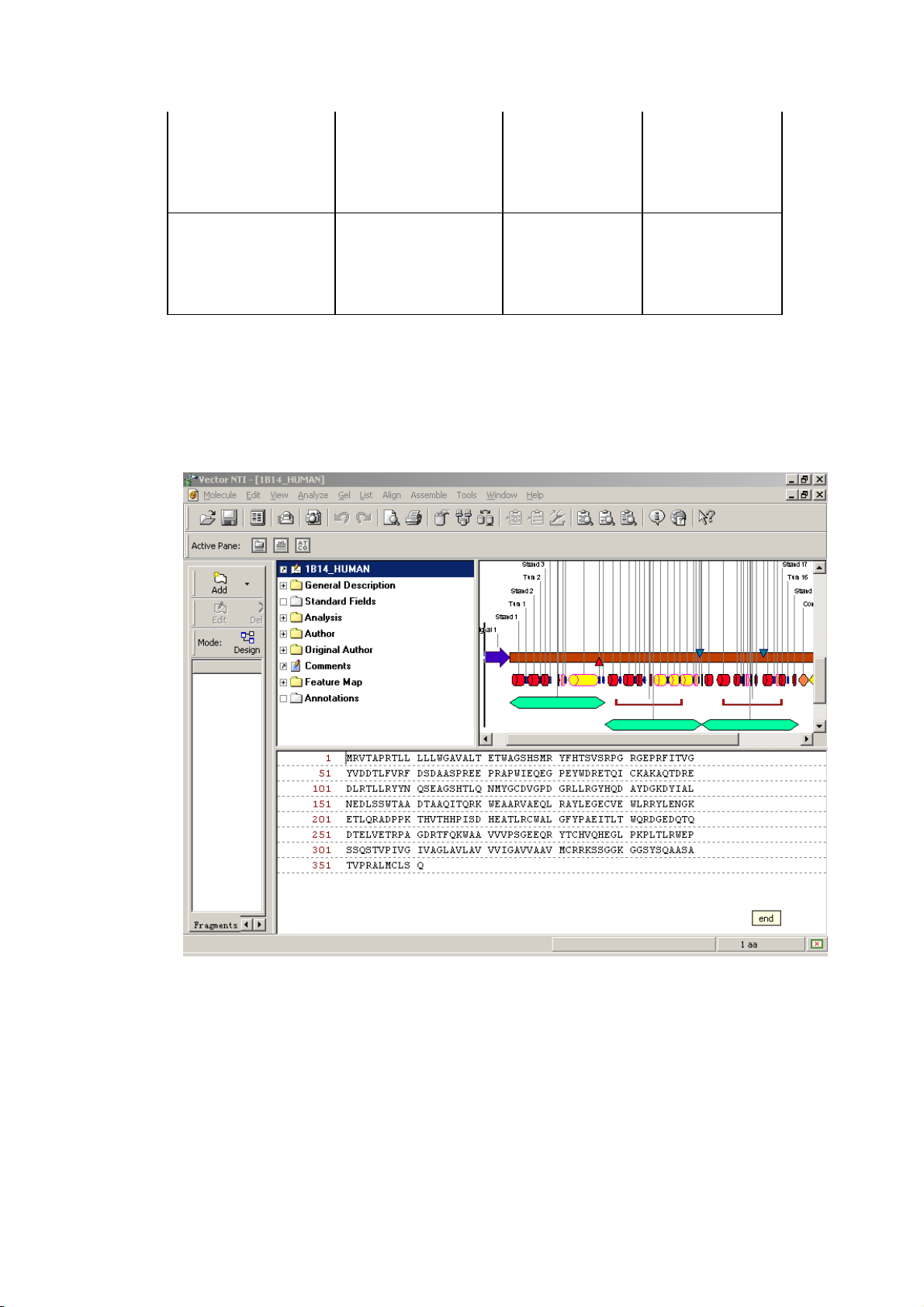
2. 爲 1B14_HUMAN 建立顯示視窗
重新回到 Database exploring——local vector NTI database 窗口,
找到 1B14_HUMAN 分子並雙擊打開。如下圖:
可以看到該蛋白分子圖形上的各種特徵顯示的十分緊湊,爲了顯
示方便起見,可以按剛才介紹的 link 命令來逐一顯示各個 feature。操
作方法和 DNA 分子一致。
關閉視窗,結束,當最後一個視窗關閉是螢幕提示 this will end
your vector NTI session,點確定(OK)關閉。
- 12 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 13
Vector NTI User Manual
第二章 Molecule Operations
目的:對 pBR322 的 general data, feature map, and sequence 進行
編輯(注意!! 蛋白質和 DNA 分子的操作是一樣的)
登錄 Vector NTI 程式
打開 pBR322 的顯示視窗(程式 vector NTIExploring local
vector NTI DatabaseDNA/RNA Molecules (MAIN)PBR322 ,雙
擊。)
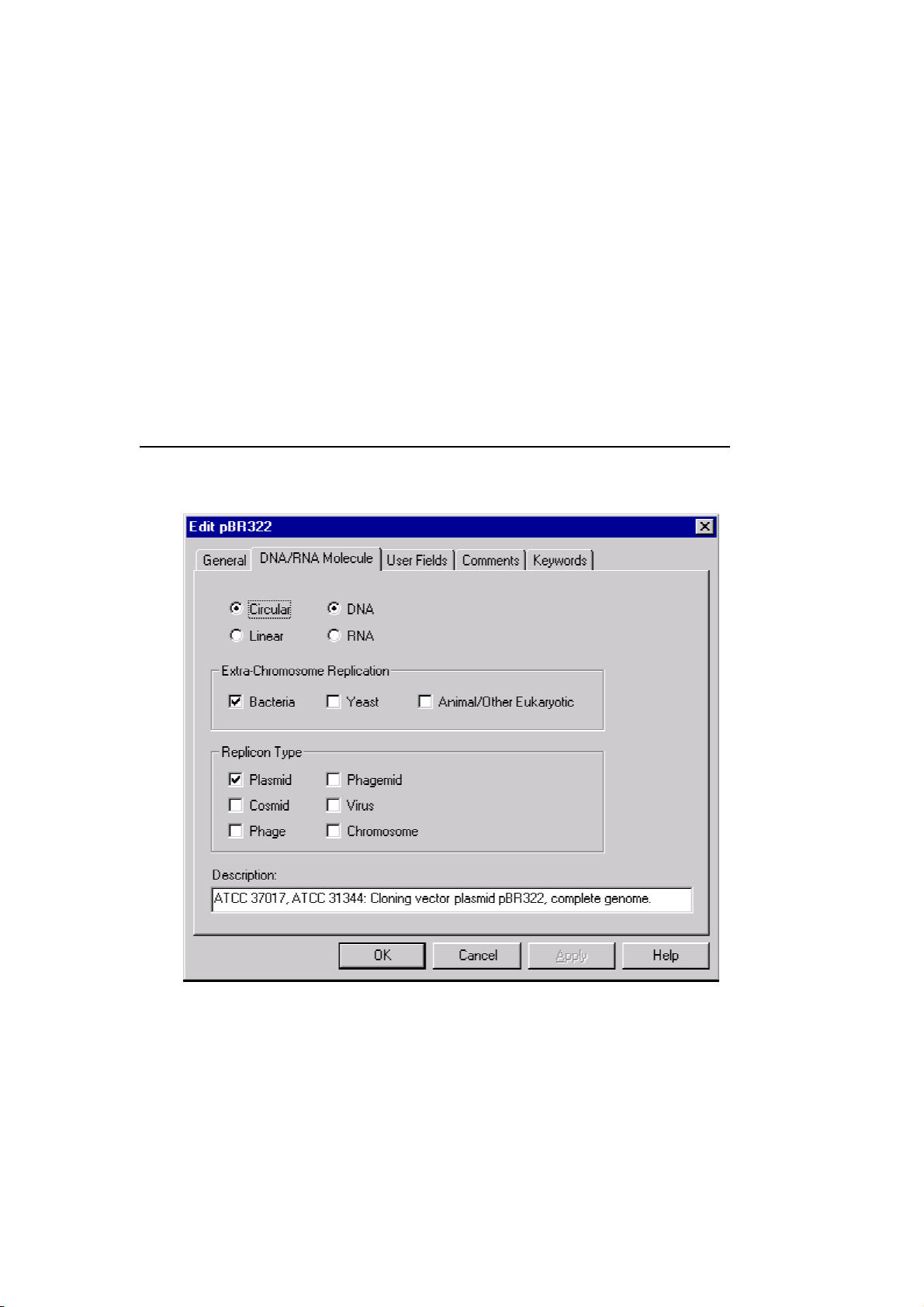
一. 對 pBR322’s 的常用資料(general data)進行編輯
在註解區的最上面,雙擊 PBR322,彈出下面窗口:
給 PBR322 加關鍵字點 keywords,在彈出的關鍵字視窗中輸入
My own plasmid,點 Add。回到 DNA/RNA Molecular 視窗,將最下
面的 description 中的內容替換成 My pBR322。點 OK(確定)。注意
螢幕的左上角 pBR322*,在 PBR322 的後面有一個星號,說明現在顯
示的是修改過的 PBR322。現在我們要在資料庫中保存這一結果:
- 13 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 14
Vector NTI User Manual
功能表 molecularsave as,在彈出的對話方塊中輸入序列的名字
My pBR322,點 OK。這時發現星號不見了,說明結果已保存到資料
庫中,這時資料庫中關於 PBR322 的 DNA 分子有兩個,一個是原始
的 PBR322(就是最初打開的那個),另一個就是我們保存的那個 my
PBR322。
二. 編輯 My pBR322’s 序列
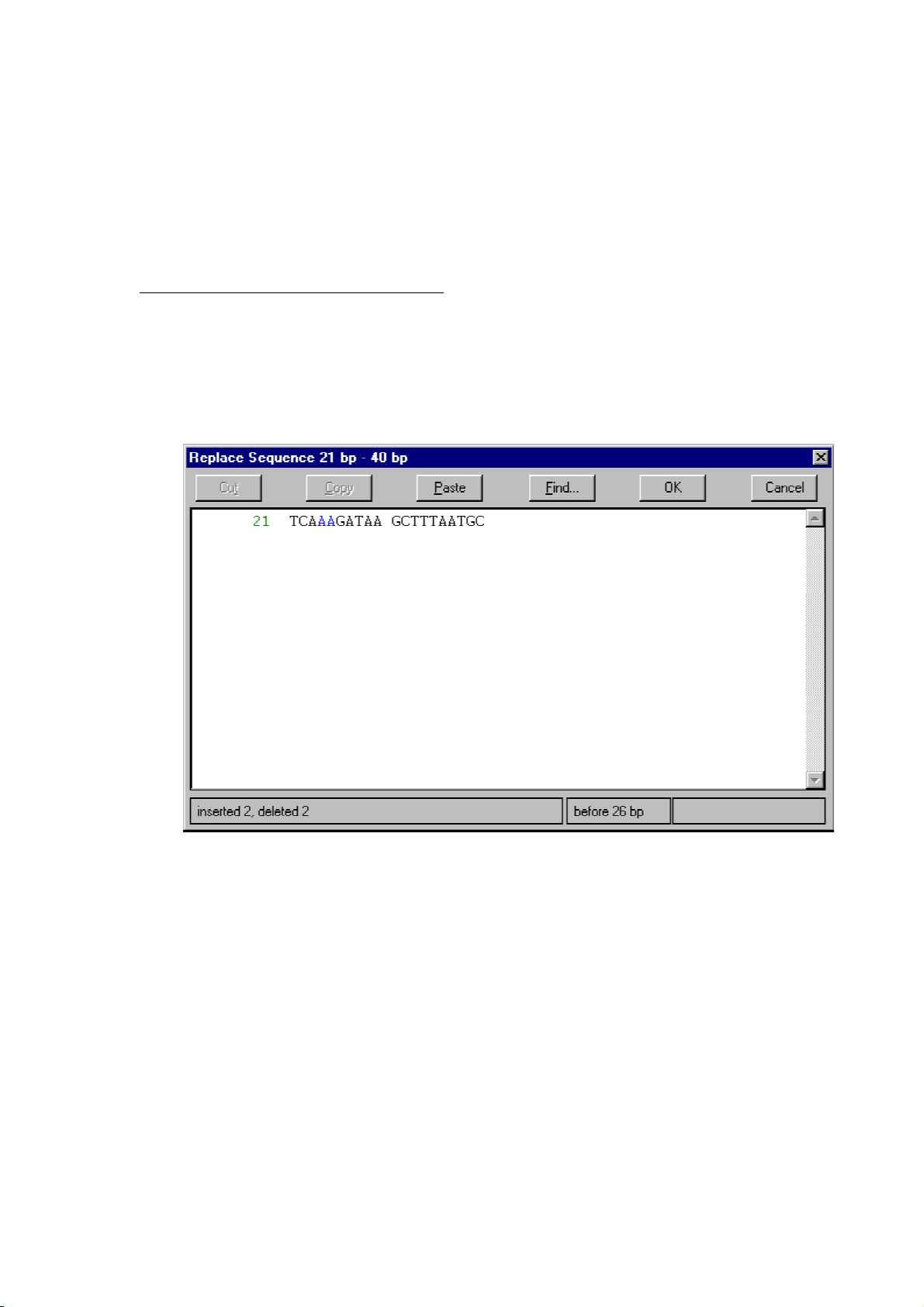
啟動序列區,功能表 editset selection,在對話方塊中輸入範圍
21-40,點 OK。會發現序列選區內含有 ClaI 和 HindIII 兩個限制酶
切位。點選功能表 editnewReplace Sequence 21 bp–40 bp,將視窗
中第 23 和 24 位的 TC 刪除分別用 AA 代替,視窗將顯示如下:
注意該視窗左下角顯示有:inserted 2,delete 2。點 OK,注意序
列中的 ClaI 位點立刻消失了。如下圖所示:
- 14 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 15

Vector NTI User Manual
將 My pBR322 的修改結果取消,不保存到資料庫
注意剛才修改完後,在螢幕左上角 My pBR322 的後面,有一個星
號,說明當前顯示的分子已經修改,下面將修改結果取消,點選功能
表 molecular——Revert To Saved,點 OK 確定。此時資料庫將剛才的
修改結果取消了。(如果需要保存修改結果則在 molecular 功能表中選
save as 命令)
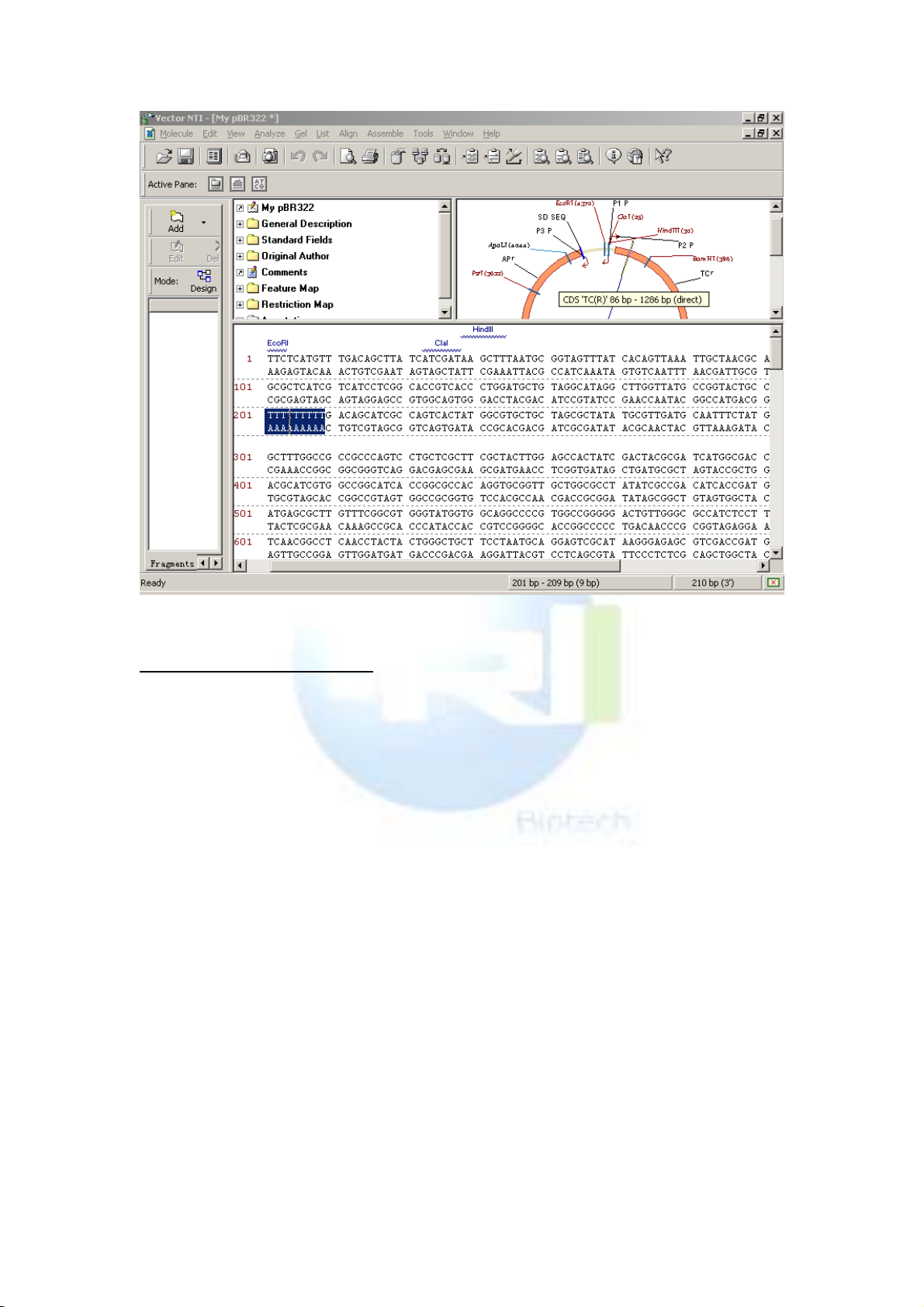
三. 插入新的序列片段
通常在編輯序列的時候需要在序列圖譜中插入一段基因或者一
段特徵序列,先找到序列中的 AP(R) 標誌( 3293 bp–4156 bp)和
TC(R) 標誌( 86 –1276 bp),功能表 editSet Caret Position,輸入
200,將游標移到 200bp 處,下面我們要在此位置處輸入 10 個 T 鹼基。
點功能表 editnewinsert sequence 200bp,在出現的對話方塊中輸
入 10 個 T,點 OK,然後會出現一個對話方塊,問你是否確認當前的
序列已經修改(CDS TC(R) is affected by sequence editing,Delete,
Delete All, Keep, and Keep All),點 keep.可以發現在 200bp 序列
處多了 10 個 T。我們將滑鼠移到 AP(R)處,可發現其位置已經順
時針移了 10 個鹼基(3303 bp–4166 bp)。其實,在原始序列中一旦插
入一段序列後,系統會自動改變圖譜中各註解的相對位置,如果插入
的序列位於某一特徵序列的內部,我們點 Keep,NTI 會自動向 3’端
移動。現在我們將滑鼠移到 TCR 處,發現其 3’端已經後移了 10 個鹼
基(1286),不過 5’端並沒有受影嚮。如下圖所示:
- 15 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 16
Vector NTI User Manual
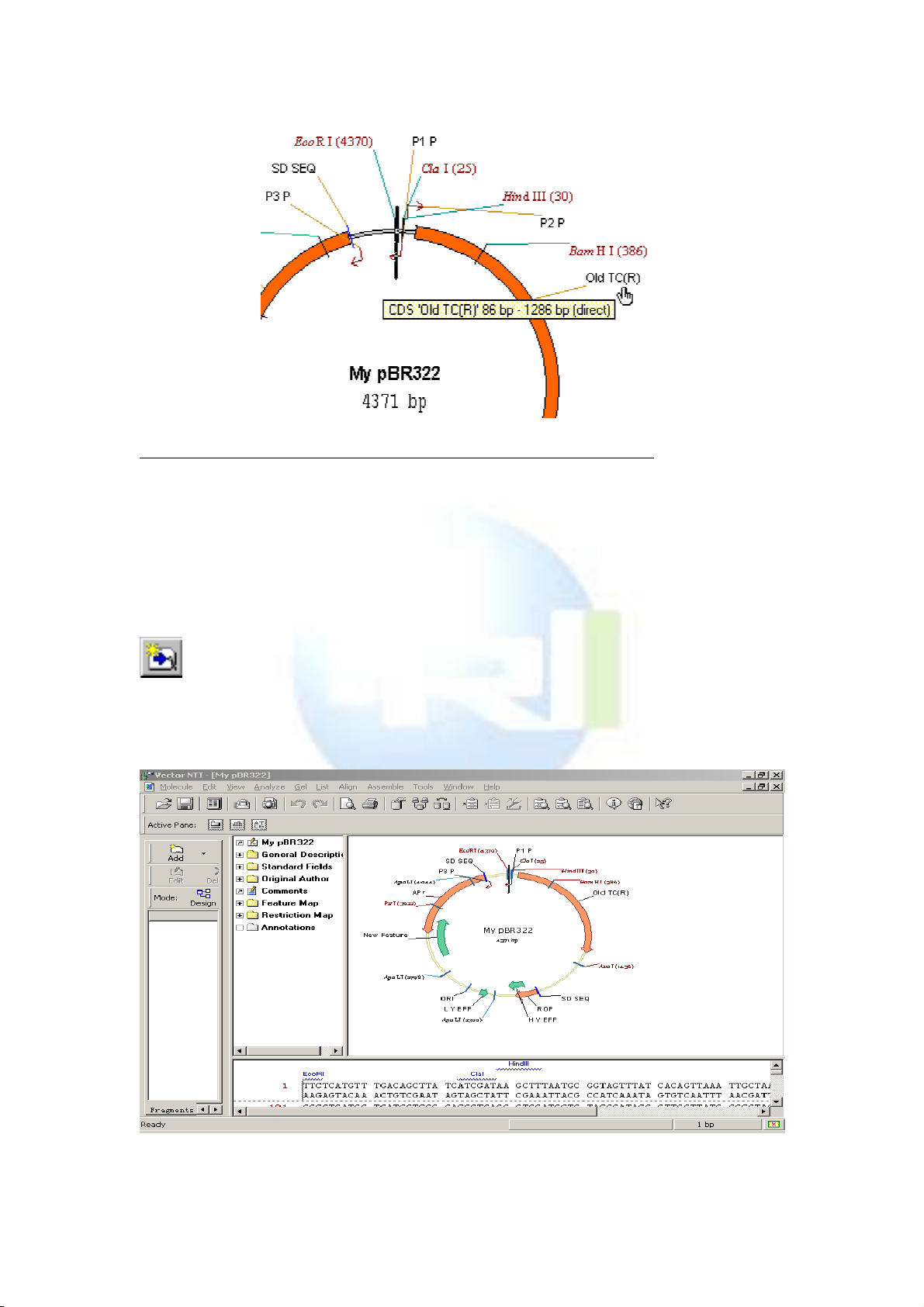
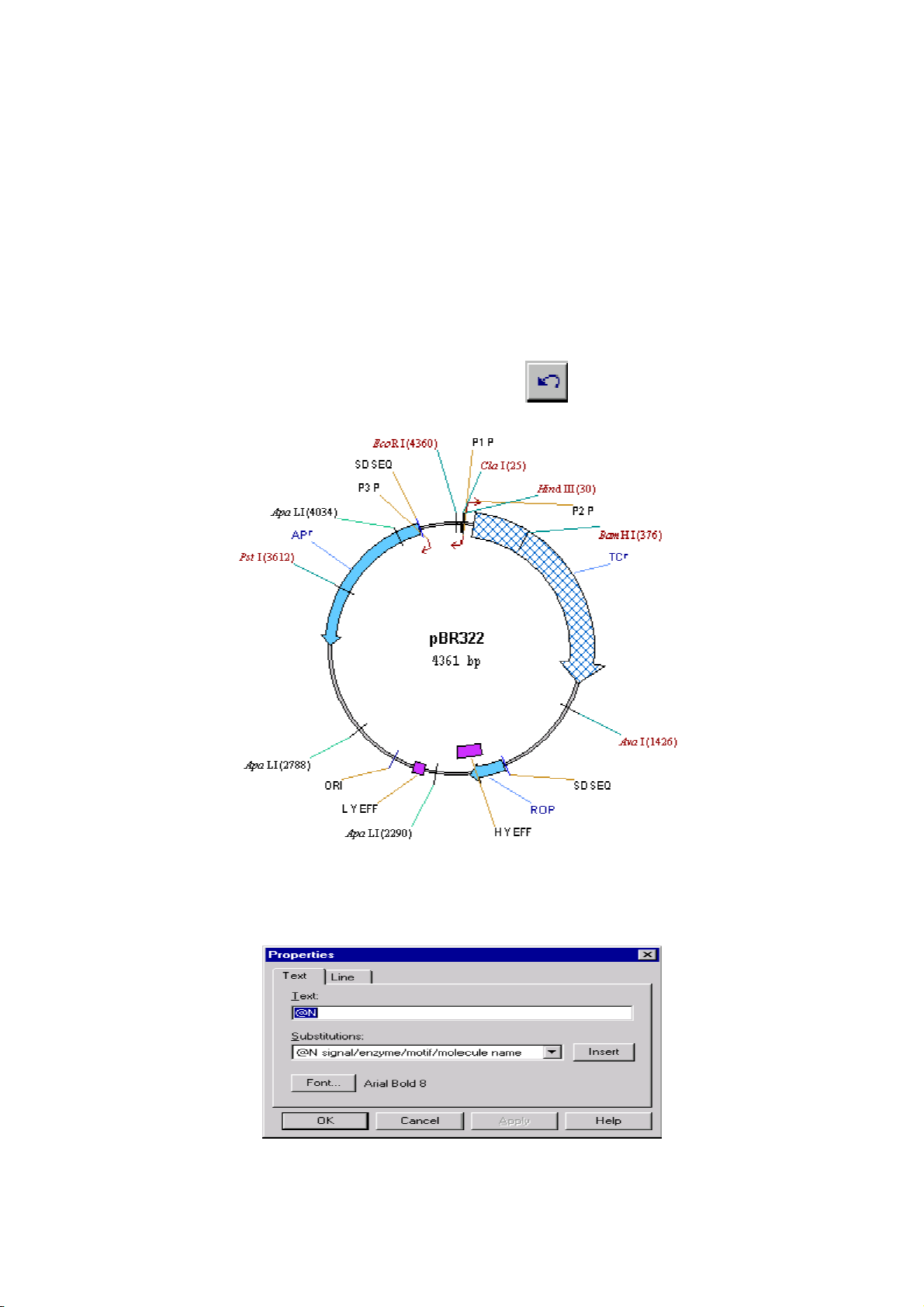
四. 編輯 TC(R) 圖示
將滑鼠移到圖形的 TCr 處,當箭頭變成“手”的形狀時雙擊(或
者點滑鼠右鍵,選 feature properties),在出現的對話方塊中使用者可
以修改 TCr 的位置、名稱和描述。我們將名稱(name)由 TC(R)改成
Old TC(R),然後在最下面的 description 中輸入描述:“10 bp fragment
inserted”,點 OK。則圖形結構變成:
- 16 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 17
Vector NTI User Manual
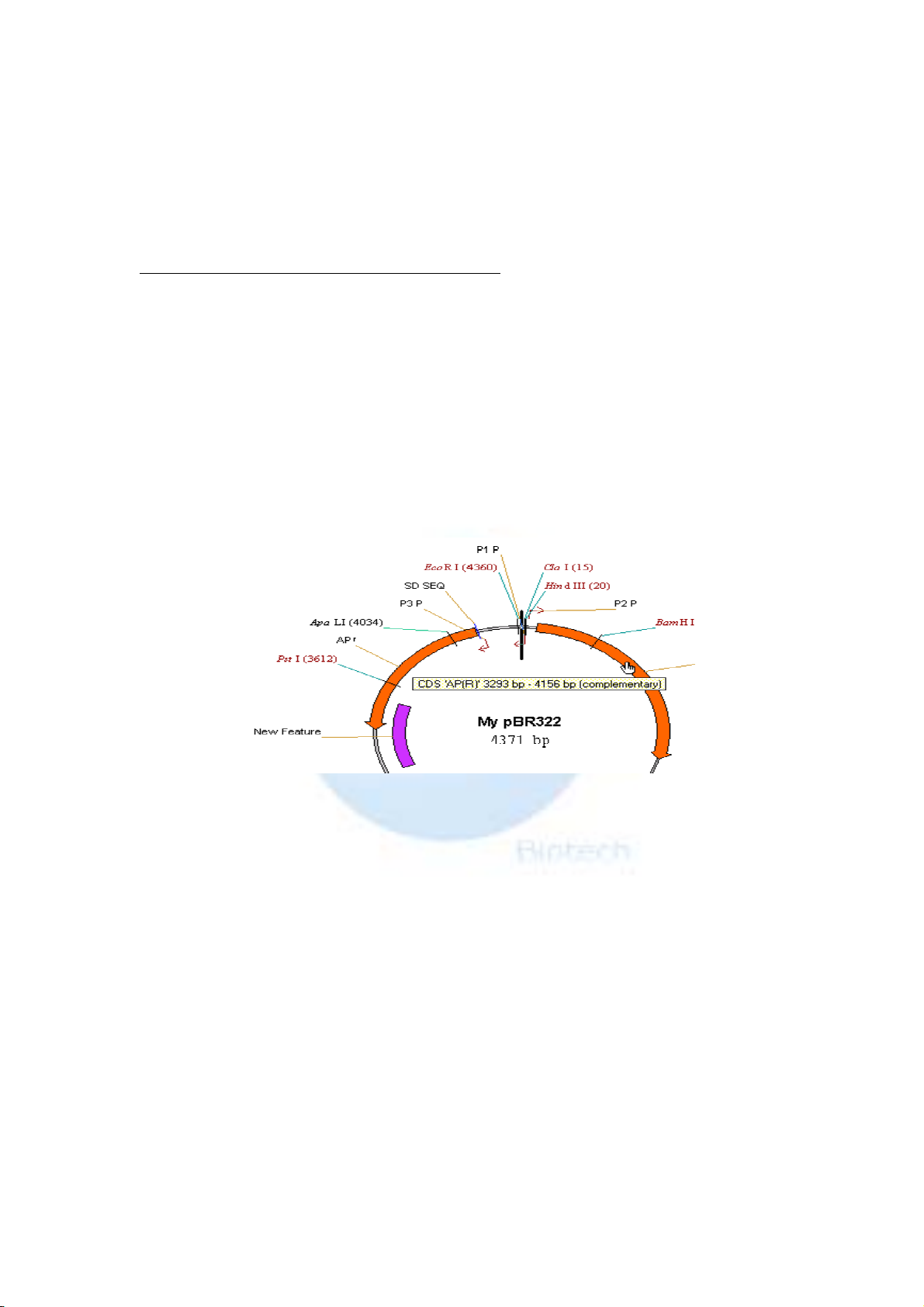
五. 刪除 P2_P 標示,並加入新的序列特徵標示
在圖譜中找到 P2P,選取,點滑鼠右鍵,選 Delete Feature From
Fmap(或者從 edit 功能表中選擇此命令),此時 NTI 將提示 P2_P will
be deleted from the feature map,點 OK(確定)。我們發現圖譜中 P2P
已經沒有了。下面我們我爲 PBR322 序列加入一個新的註解:
editset selection輸入 3000-3500bp,點 OK。在工具列中找到
(add features)按鈕,點它。(也可以從 editnewadd feature to
FMap 進入)。在出現的對話方塊 feature name 中輸入 New Feature,
注意 NTI 原始設定的特徵類型(feature type)爲 Misc. Feature,使用
者可以從列表中選擇自己認可的特徵。最後點 OK 。如圖:
- 17 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 18
Vector NTI User Manual
下面將 My pBR322 的修改結果保存到資料庫中:功能表
molecular——save as,(如果不想改名的話)點 OK,NTI 將提示 My
pBR322 已經存在,是否覆蓋,點 overwrite.
六. 修改 My pBR322 的起始座標
這樣可使剛才插入的 10bp 片段和最初的PBR322 具有一致的坐標
系。功能表 MoleculeoperationsAdvanced (DNA/RNA)Change
Starting Coordinate。在出現的對話方塊 new start(新的起始點位置)輸
入:11(因爲剛才插入了 10bp,所以起始點是 1+10=11)。點 OK,
此時 NTI 會提示當前座標已被修改,是否繼續,點 OK 確定。現在我
們會發現顯示視窗中 TCR 的位置已經變成了(76bp–1276bp),還有
AP(R)的位置(3293 bp–4156 bp),這都和最初的 PBR322 一致。
如下圖:
退出顯示視窗,關閉 NTI
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
- 18 -
Page 19
Vector NTI User Manual
第三章 Graphical Representation
目的:以 DNA 分子爲例,對分子圖形的展示進行操作(蛋白質
的操作和 DNA 類似),爲 pBR322 圖形建立一個展示視窗,並保存到
分子註解檔中
一. 爲 pBR322 圖形建立一個展示視窗
1. 登錄 Vector NTI 程式
2. 通過新視窗打開 PBR322(與前兩章的打開方式略有不同),
在 NTI 主窗口中,點選工具列最左邊的打開文件夾(或者
molecular——Open),在彈出的 Open 窗口中點選 database
DNA/RNAs,在列表中找到 PBR322,然後 OK。
3. 顯示視窗的調整,比如我們想觀察圖像的詳細情況,首先用尺
規拖動圖形視窗,至於序列和註解區,可以很小,因爲我們
的目的是看圖。然後點
或 讓圖形放大或縮小,直到滿
意爲止,如果使用者想一步步的看放大效果,可以按住 shift
的同時點
。
二. 修改圖形的自動排列設置
按住 ctrl 的同時,點 (Standard Arrangement),使用者可以
根據彈出的小功能表,來選擇圖形線條的粗細和字體的大小,直到滿
意爲止。
三. 修改圖形的編碼區信號( CDS signals)設置
點中圖形中任意編碼區(粗箭頭),然後按滑鼠右鍵,在彈出的
下拉功能表中,選 CDS Display Setup,使用者可以在彈出的 Graphics
Display Setup 對話方塊中修改所選編碼區的名稱、顔色標記、箭頭的
粗細等,(實際上 NTI 默認的就很好了)。使用者也可以通過點 more
來進行更多的修改(比如字體和大小等,和 word 中字體的處理很相
似),修改完後一路 OK 點下去即可。注意這種修改只是針對本窗口
中的分子,對其他分子沒有影響。
- 19 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 20
Vector NTI User Manual
比如我們將箭頭填充成蘭色。下面我們保存這一設置:點選
(display setup)右側的小三角形符號,在彈出的功能表中選 Save
Settings As,然後在彈出的視窗中給剛才的設置風格取個名字,不妨
叫 blue,點 OK,注意此時 NTI 會提示是否保存其他沒用過的風格,
點 NO。現在再點選
(display setup)右側的小三角形符號我們會
發現 blue 已經在下拉功能表上了。
四. 打開圖片編輯方式
NTI 提供了兩種編輯圖片的方式,分子編輯方式 (原始設定)和圖
片編輯方式。啟動圖形區然後按
(edit picture)按鈕。然後點中
圖形中任意標記或者箭頭,按住滑鼠右鍵,在彈出的下拉功能表中選
properties(屬性)或者 style(風格)等,進行設置,注意此時設置的僅僅
是所選的箭頭或者標記,而不是整個分子(在之前所設的是整個分
子,請注意比較,兩種方式的轉換可通過
按鈕進行)。
五. 修改 TC(R)圖形
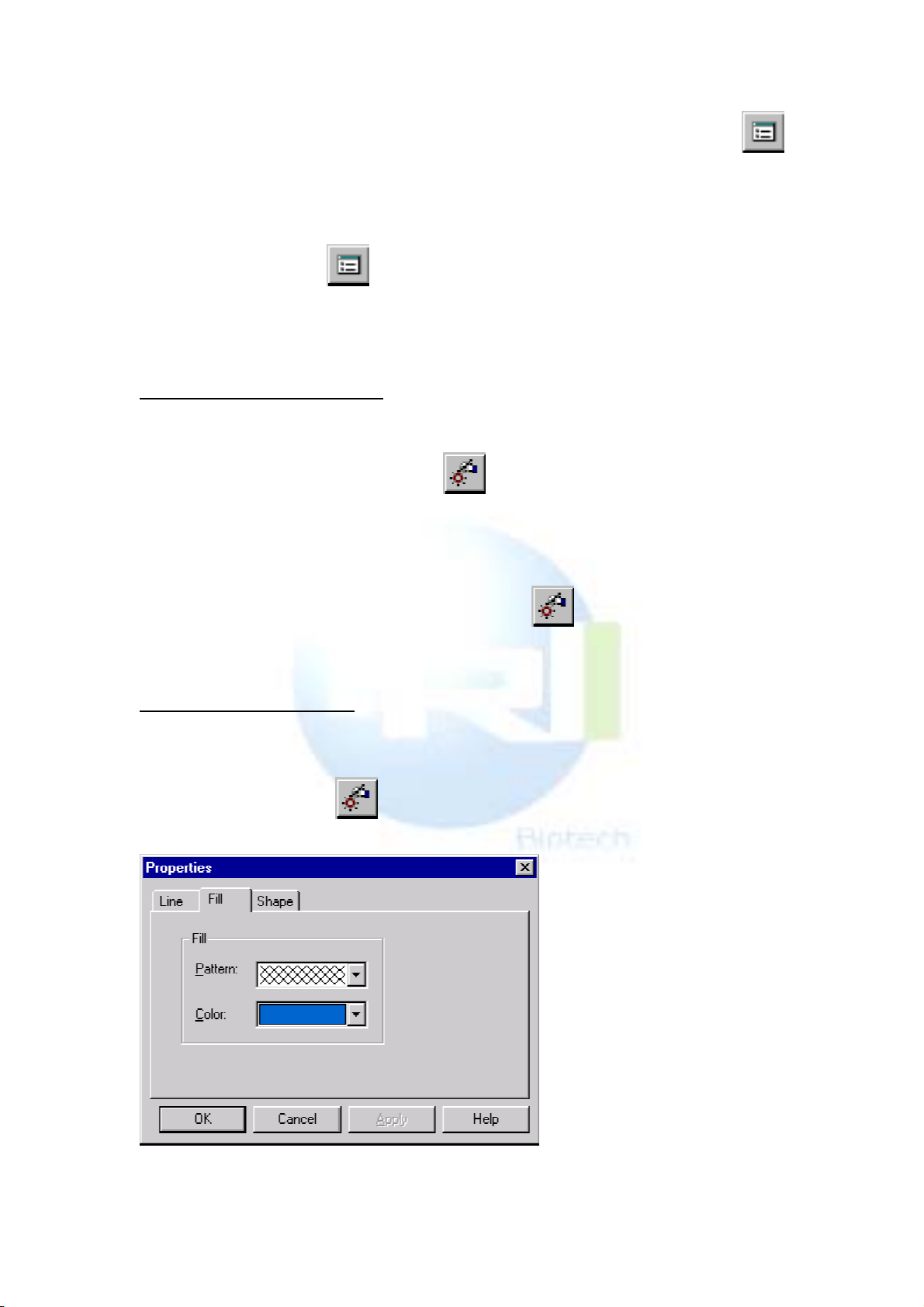
1. 將 TC(R)箭頭變成蘭色網格
操作如下:點中
按鈕,點中 TCR 箭頭,按住滑鼠右鍵,選
propertiesfill,按右圖方式選擇:
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
- 20 -
Page 21
Vector NTI User Manual
點 OK
2. 放大 TC(R)箭頭
點中 TCR 後,當滑鼠在箭頭附近移動時,會出現兩種十字形標記:
和 ,通過滑鼠拖動 可以改變箭頭的胖瘦,而拖動 則可以改
變箭頭的相對位置(移到圓內或圓外)。如果想讓箭頭按圓圈轉動,
可以在按住 ctrl+shift 同時,拖動
。注意這種拖動並不能修改分子
本身,僅僅是改變位置而已,如果想修改分子中的標記,請使用第二
章介紹的方法。若想取消可以按
取消,如下圖:
3. 修改 TC(R)標記的格式
將滑鼠移到 TCr 標記上,雙擊。顯示下面的對話方塊(就是屬性)
- 21 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 22

Vector NTI User Manual
點 Font,選擇斜體 18 號字,點 OK。TCR 將顯示如下圖:
我們發現,字體大了。點 (Standard
Arrangement)按鈕,使標記的信號回到標準位置,如果圖片已經修
改,NTI 還會不厭其煩的提示是否繼續,點確定吧。
六. 加入註解注釋
看到視窗工具列最右側的迴紋針符號了沒,點它(如果沒反應,
點
後再點迴紋針符號),在彈出的對話方塊中輸入“Clone into
pUC19”,點 OK。此時加入的注釋出現在圓圈的中央,用滑鼠拖到
TCr 的下面即可(可點滑鼠右鍵從 properties 中選擇字體的大小和顔
色)。如下圖:
也可以先選取剛才添加的註解,點功能表 editDelete Annotation
進行刪除。注意以上修改的僅僅是顯示的介面,NTI 並不能將修改的
顯示結果保存到資料庫中,所以使用者也可以發現在左上角並沒有*
號標記,也就是資料庫中的分子沒有改變,如果想讓它改變的話,必
須將之存到資料庫中。
七. 將 pBR322 分子顯示結果保存到文檔文件中
功能表 Moleculesave assave as file,名稱 NTI 已經給起好了,
- 22 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 23

Vector NTI User Manual
就是 pBR322.gb。選擇好要保存的目錄,點 OK。如果希望存剛才建
立的 blue,可以在保存前選擇
右側的三角按鈕選擇 blue。此時系
統會提示是否建立 html 文件描述分子,點 yes(這樣我們就可以和朋
友通過 internet 進行交流了)。注意該文件檔完全與資料庫相關聯,其
顯示的資訊與資料庫中的資訊一致。但風格與資料庫不同,比如箭頭
的顔色、字體的大小等(當然由使用者決定)。現在使用者可以先關
閉文件檔然後再打開就可以發現顯示的風格和資料庫中的風格完全
不同(不過內容還是一樣的)。注意:對於原先資料庫中沒有的分子
(比如使用者根據自己的需要自己構建或從 GENBANK 下載的,並
不能直接修改顯示風格,如果想修改其顯示風格,先將該分子保存到
資料庫中,這樣就可以了)
退出程式
- 23 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 24

Vector NTI User Manual
第四章 BLAST Search
一. 簡介
BLAST(Basic Local Alignment Search Tool) 是以一段 DNA 或蛋
白質序列來針對現有的 DNA 或蛋白質序列資料庫來進行搜尋比對的
一種方法,在 Vector NTI 中是以 internet 連接NCBI (National Center for
Biotechnology Information)來使用 NCBI 的BLAST 程式及序列資料庫
來進行比對,NCBI 的 Genbank 所收集的是目前最完整的公用(public
domain)DNA 及蛋白質序列資料庫,透過 BLAST 比對可以找尋資料庫
中與query 序列具同源性(homology)的序列,接下來可依此利用AlignX
進行多序列並列分析。使用者也可透過架設自有的 BLAST SERVER
來以 Vector NTI 比對私人序列資料庫。
二. BLAST 搜尋視窗
Ve ct or N TI 預設的 BLAST search engine 是位於 NCBI website
(
http://www.ncbi.nlm.nih.gov/blast/blast.cgi)。使用者可由開始程式集來開啟
BLAST Search 視窗。如下圖:
另一種更方便的方式,是在 Vector NTI 裏面,直接選擇想要比對的
序列,可能的選擇方式很多, 例如直接點圖形中的某個 CDS, 再由
ToolsBLAST Search 即可開啟視窗,如下
- 24 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 25

Vector NTI User Manual
這就表示只有所選的序列會被用來進行 BLAST Search,而且等一
下會自動將序列貼入,sequence page 中。
接下來會出現以下視窗:
按 OK, 會出現下列視窗:
預設的 search engine 是 BLAST 2.0, 接下來可依需求選擇
Program 和要比對的資料庫::
- 25 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 26

Vector NTI User Manual
D
D
以
P
以
D
P
以
P
以
資料庫。
D
D
將
Application Query Database Description
blastn
blastp
blastx
tblastn
tblastx
NA
rotein Protein
NA
rotein DNA
NA
Menu Identifier
nr
EST(+ human,
mouse, others)
NA
DNA 搜尋 DNA 資料庫。
protein 搜尋 protein 資料庫。
rotein
DNA 搜尋 protein 資料庫。
protein 搜尋 translated DNA
NA
DNA 轉譯成六個reading frame 搜尋
轉譯成六個reading frame 的 DNA 資料
庫。
Description
All non-redundant GenBank sequences and CDS
translations
EST (Expressed Sequence Tags)
Others includes all organisms except mouse and
human
GSS
HTGS
PAT
Yeast
Mito
vector
E. coli
month
Genome Survey Sequence, includes single-pass
genomic data, exon-trapped sequences, and Alu
PCR sequences
High Throughput Genomic Sequences
Protein sequences from the Patent division of
GenBank
Saccharomyces cerevisiae protein sequences—a
database of sequence fragments of the Yeast
complete genome
Mitochondrial DNA sequences
Vector subbase of GenBank
Escherichia.coli genomic CDS translations
All new or revised GenBank CDS translation +
PDB + SwissProt + PIR + PRF released in the
last 30 days
- 26 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 27

Vector NTI User Manual
在 Sequence Page 使用者可以 key in 或 copy/paste 想要比對的序
列或 select Search Open 來打開要比對的序列檔。
在 Parameters Page 可以用來設定搜尋的 parameter。
Expect 值是用來限制所找的到序列的相似程度,若希望找到更多
相似度較低的序列可以將它提高, 不過最好是使用預設值。
Matrix Page 可以容許使用者選擇不用的 matrix table,這通常在比
對蛋白質序列時才有需要,在此還是建議使用預設值
BLAST Options—可以選 View Options 更改 option,例如希望
display 的序列數目,再按 OK
- 27 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 28

Vector NTI User Manual
回到 BLAST Search Viewer, 開始 BLAST search.
三. BLAST Search Results
可以直接由 BLAST Search Viewer 下方觀看 BLAST 是否已結束
搜尋,再雙擊打開 BLAST Viewer。或是由開始 功能表Vec t or
NTIBLAST Viewer 打開來看 BLAST 所得的結果,可以按
結果, 選取要看的結果將它打開後可看到下面的視窗
來尋找
在畫面左方是 BLAST Viewer 的 Text Pane,裏面包含整個搜尋的
- 28 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 29

Vector NTI User Manual
Query 及結果的整體說明,在 Hit 下面就是比對所得的所有結果,以上
圖為例,資料庫中共有 16 條序列與 query 具 homology。可以選擇其中
一個序列,雙擊檔案夾就會出現關於這條序列與 query 比對所得的文
字說明。若選擇其中的 hyperlink,會自動將這條序列由 NCBI 下載下
來並且直接用 Vector NTI 打開來。
在右方就是這條序列與 query 序列的比對情形以圖形來顯示. 最
上方是兩條序列間比對的相似程度的情形,中間的則是以整體來
看,Hit 的序列與 query 序列之間,相似的區域的位置關係,下方則是以
所有Hit 到的序列的位置而言(藍色),這條序列(紅色)與query及其他序
列間的位置關係。最下方的視窗則是 query 及所選擇的序列之間並列
分析的情形。
四. Saving BLAST Search Results
要存下BLAST search 的結果,選 Blast Results Save As 或按 ,.
結果可以存成 Vector NTI database 或是 BLAST result file,如下圖:
選擇要存的目錄及檔名,再按 OK 即可。
- 29 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 30

Vector NTI User Manual
- 30 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 31

Vector NTI User Manual
第五章 AlignX
一. 開啟與執行:
如圖於NTI 之Database Exploer 中選取所要之序列點選上方之
AlignAlignX-Align Selected Molecules
VNTI 會開啟 AlignX 視窗並載入所選取之序列,如下圖:
- 31 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 32

Vector NTI User Manual
選取所要進行分析之序列 點選上方之 AlignmentAlign
Selected Sequences
程式運算時間端視於所給予之工作及電腦之配備,結果如下圖:
結果包含演化樹圖,序列分析圖,多重序列比對圖。
- 32 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 33

Phylogenetic Tree
Graphics
Alignment
Vector NTI User Manual
開啟 Similarity Table 與 Dot Matrix Plot:
選取 AlignmentShow Similarity Table 或 Show Dot Matrix Plot
- 33 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 34

Vector NTI User Manual
選換分析方法:此功能為將 BioPlot 與 AlignX 結合,使 AlignX
的結果可進行 BioPlot 的分析。
於 Alignment Pane 範圍View List of Analysis
選取分析
方法。
- 34 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 35

Vector NTI User Manual
二. 參數設定與調整:
1. Alignment 基本設定:
選取 AlignmentAlignment Setup
- 35 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 36

Vector NTI User Manual
可依據需要更改設定,詳細資料可點選說明鍵,原廠提供之說明。
2. Alignment Display Setup:
滑鼠雙擊 Alignment Pane 之範圍會出現 Alignment Display Setup
- 36 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 37

Vector NTI User Manual
3. Plot Setup:
於 Alignment Pane 之範圍內,按滑鼠右鍵出現:
出現的內容依據你滑鼠當時點選哪一個分析圖,可以更改該圖形
格式及顏色。
- 37 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 38

Vector NTI User Manual
Alignment Pane 中序列之排列順序可自由上下調整:
以滑鼠點選序列,按住左鍵,可上下移動至所選之位置。
- 38 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 39

Vector NTI User Manual
Edit Alignment:可手動調整 Alignment 結果
於 Alignment Pane 之範圍View Edit Alignment
三. 資料輸出與列印
1. Alignment 資料輸出:
a.於 Alignment Pane 範圍 ProjectPrint 此為程式內建之列印格式。
b.於 Alignment Pane 範圍EditCameraFormat 選取 Text 為文字輸出。
Forma 選取 Metafile 為圖形輸出。
Camera
Word ,PowerPoint 等進行編輯。
之功能為將資料暫存於剪貼簿中,可開啟熟悉之程式如
程式畫面上之所有圖形及資料皆可以 Camera 之功能將資料輸出
編輯。資料輸出之範圍以該選取之 Pane 為主。
Phylogenetic Tree 之數值輸出:
於 Phylogenetic Tree Pane 之範圍ViewExport Phylogenetic
TreeSave File。
此檔案可以記事本開啟。
- 39 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 40

Vector NTI User Manual
第六章 AlignX-Blocks
(此功能只適用於蛋白質序列分析)
一. 開啟與執行:
1. 如圖於 NTI 之 Database Exploer 中選取所要之序列點選
上方之 AlignAlignX Blocks-Align Selected Molecules
2. VNTI 會開啟 AlignX Blocks 視窗並載入所選取之序列,如
下圖:
- 40 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 41

Vector NTI User Manual
3. 選取所要進行分析之序列點選上方之 BlocksSearch for
Blocks
程式執行時,如下圖:
4. 程式運算時間端視於所給予之工作及電腦之配備,呈現之
結果如下圖:
- 41 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 42

Vector NTI User Manual
Text
Schematic
Block List
Alignment
二. 參數設定與調整:
1. AlignX Blocks 基本設定:
選取 BlocksAlignX Blocks Setup
可依據需要更改設定,詳細資料點選說明鍵,看原廠提供之
說明。
2. Edit Block:可命名 Block 及更改 Block 顏色。
- 42 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 43

Vector NTI User Manual
於 Block List Pane 之範圍,選取 BlocksEdit Block
3. Edit Color:更改 Text 的顏色。
於 Alignment Pane 之範圍內,選取 ViewEdit Color
4. Link Setup:將位於不同位置之相類似 Blocks 並列比較分
析。
於 Block List Pane 之範圍,選取 BlocksLink
- 43 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 44

Vector NTI User Manual
三. 資料輸出與列印
1. Alignment 資料輸出:
a.於 Alignment Pane 之範圍 ProjectPrint 此為程式內建之列印
格式。
b.於 Alignment Pane 之範圍EditCamera Format選取Text 為文字
輸出。
Format 選取 Metafile 為
圖形輸出。
Camera
之功能為將資料暫存於剪貼簿中,可開啟熟悉之程
式如 Word,PowerPoint 等進行編輯。
2. 程式畫面上之所有圖形及資料皆可以 Camera 之功能將資
料輸出編輯。資料輸出之範圍以該選取之 Pane 為主。
- 44 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 45

Vector NTI User Manual
第七章 BioPlot
一. 開啟與執行:
1. 如圖於 Vect or N TI 圖形中之中選取所要分析之序列點選上方
之 AnalyzeBioPlot-Analyze Selected Molecule
2. VNTI 會開啟BioPlot 視窗並載入及分析該序列,如下圖:
Text
Plot
Sequence
- 45 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 46

Vector NTI User Manual
3. 檢視序列之一般分析結果於 Text Pane 中點選
果如圖:
結
4. Broadcast Selection:可將選取之序列同步呈現於不同之程式中,
如 Vector NTI。選取序列View Broadcast Selection 結果如
圖:
- 46 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 47

Vector NTI User Manual
5. 更改及增加新的分析方法(Analysis List):
選取 AnalyzeAnalysis List
種,Protein 五十種)。
選取所需之分析方法(DNA 八
6. BioPlot 基本設定:
選取 AnalyzeBioPlot Setup
可設定序列之排列方式
可設定欲顯示之 Feature
- 47 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 48

Vector NTI User Manual
可依據需要更改設定,詳細資料可看原廠提供之說明。
7. Plot Display Setup:
滑鼠雙擊 Plot Pane 中,各分析圖之範圍會出現該分析方法之內
容,如圖,出現的內容依據你滑鼠當時點選哪一個分析圖,可以更改
該圖形格式及顏色。
8. Plot Overlapping:疊合多個分析圖。
. 於 Plot Pane 之範圍 ViewEdit Layout
Overlapping
可以滑鼠自由移動圖形。
- 48 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Enable
Page 49

二. 資料輸出與列印
Vector NTI User Manual
1. BioPlot 資料輸出:
a.於各 Pane 之範圍 FilePrint 此為程式內建之列印格式。
b.於各 Pane 之範圍EditCamera
Camera 之功能為將資料暫存於剪貼簿中,可開啟熟悉之程式
如 Wo rd,PowerPoint 等進行編輯。
2. 程式畫面上之所有圖形及資料皆可以 Camera 之功能將
資料輸出編輯。資料輸出之範圍以該選取之 Pane 為主。
- 49 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 50

Vector NTI User Manual
第八章 ContigExpress
一. Introduction
ContigExpress 用於小片段序列串連,可以將純粹文字檔或是由
automatic sequencer 所輸出的檔案皆可直接進行分析;而串接後的片
段稱為 “contigs”.
所有”contigs”皆可直接於 Vector NTI 中開啟,並可透過工具列中
的”BLAST Search”將 data 上傳至 WWW 中進行比對。
This tutorial can be completed in one session or divided into three
sessions as noted. At the end of this tutorial, you will be able to:
Create a ContigExpress project
Navigate and manipulate the CE Project Explorer window
Assemble contigs
Edit fragments and contigs in the Fragment Window or the Contig Window
Follow the steps of the tutorial in the order shown. Figures show what your
screen should look like at various points along the way.
二. Project Explorer
1. Add fragments to the project
輸入檔案的方法可以分為數種:
從檔案中輸入時,可以從 Ad
d Fragments 此時會出現如此畫面
- 50 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 51

Vector NTI User Manual
此時可依據檔案的格式選擇;例如我們選擇”From ABI file”
Fig. 17. 1
Note: Sometimes the names of the imported fragments do not
directly correspond to their original file names – ContigExpress assigns
names that can be found inside the imported files to the newly imported
fragments. (In this example, Sample1303.abi becomes xb-control on the
list).
2. Examine the CE Project Explorer window.
當序列輸入後,即可出現如此畫面:
- 51 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 52

Fig. 17. 2
Vector NTI User Manual
3. Create Assemblies of Contigs
選擇串接方法,在此提供兩種分析方法:
Algorithm
- 52 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 53

Vector NTI User Manual
Pairwise
Assembly
Linear
Assembly
best for assembling ten or fewer
fragments
faster initialization
lower memory use
may be faster for small numbers of
fragments
more stringent default conditions
user can set more assembly
parameters; allows higher degree of
assembly customization
best for assembling 11 or more
fragments
slower initialization
higher memory use
faster assembly of large number of
fragments
less stringent default conditions;
may allow assembly of fragments
with smaller regions of overlap
fewer parameters to set; only
Minimum length of any overlap
parameter is used
利用滑鼠可選擇要串接的序列或是全選全部分析。 點選快速鍵
).
中的 (
注意:至少要兩個以上的序列採可進行分析。
.
.
分析完後會將分析的結果呈現如下圖;”Contig 1”代表新串連的
片段。將滑鼠點選”contig 1”此時會出現如
- 53 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 54

Vector NTI User Manual
此時可將不同片段的”峰圖”做比較。
三. Working in Fragment Window
1. Open and inspect the Fragment Window
- 54 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 55

Vector NTI User Manual
Text Pane
)
當打開文字說明視窗(
時,General Description 代表此序列
的基本資料,可以透過編輯給予不同的描述。Chromatogram Data 代
表 data 從 Sequencer 輸出時的日期與時間,是不可變動的資料。
Sequence pane
當打開序列視窗按鈕時 (
與雙股轉換鍵(
), 呈現不同的序列表達方式:
)僅會出現序列視窗;可以透過單鼓
- 55 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 56

Vector NTI User Manual
.
2. Sequence selection techniques
In ContigExpress 用戶在點選序列時,於視窗的右下方會出現序
列相對位置的訊息。
Position box
Selection box
也可透過”selection box”直接輸入所要選取的序列或區域。:
Click on the Selection box on the Status Bar, opening the Set
- 56 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 57

Vector NTI User Manual
Selection dialog box. Set the selection to 10 bp – 30 bp and press OK:
The selection is highlighted in the Sequence Pane and shown in the
Selection Box on the status bar:
- 57 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 58

Vector NTI User Manual
3. Translate the nucleotide sequence
選取一段序列進行轉譯的分析,用戶可利用快速鍵: ( ) 做正向
轉譯;或是藉由 (
). 做反向反意轉譯:
點選 (
) 進行單字與三字氨基酸呈現轉換;利用( ) [an
“eraser”]. 清除不要分析的部分。.
點選(
)可就序列進行 ORF 預測,如下圖:
- 58 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 59

Vector NTI User Manual
4. Edit the nucleotide sequence in the Sequence Pane
若用戶將序列進行編輯時,如果執行”DELETE”指令時,被刪除
的序列處戶出現 (↓) 以示區別:
此時的序列本身會往前計算,但在 Chromatogram data 中,原始
的紀錄會被保存,但序列會被以(-)來省略。.
相對地,若是自行加入序列的話會再加入的地方出現(↑)來標
記,在 Chromatogram data 中,此區域的峰圖會出現空白的”gap”
假如我們以取代的方式修改序列,如“GG”被輸入以取代 “AA”,
此時原本的序列會保留於下方,而峰圖也會將原始數據保留但序列做
更動。:
- 59 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 60

Vector NTI User Manual
5. Analysis the Sequence via BLAST
藉由視窗上方工具列中的>Tools>BLAST search, 此時會出現一
個對話方塊
點選”OK”,這時回直接把序列輸入至”BLAST search”功能中,
即可進行搜尋的工作。
四. Working in the Contig Window
Selecting and opening a fragment brings up Fragment Window.
Selecting and opening a contig, opens a Contig Window.
1. Open and examine a Contig Window.
- 60 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 61

Vector NTI User Manual
藉由開啟一個”Contig”檔案瞭解各個視窗提供的訊息所代表的意
義。
Graphics Pane
Click the Contig Graph button (
) in the Window toolbar to
activate the Graphics Pane.
The Graphics Pane contains horizontal arrows representing the
relative positions of the fragments forming the contig. The arrowheads
indicate the direct or complementary position of the respective fragment.
在下圖中的直線於第 270 base 出現一個”gap”,這代表進行
assembling 時有序列出現不”match”現象,這個訊息可作為 quality
control 的依據。.
- 61 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 62

Vector NTI User Manual
2. Review the contig in Contig Alignment Pane
點選 (
) 顯示各片段排列情形:
The fragments names list contains all fragments used in this
contig
The scale gives a reference for all fragments and the consensus
shown in the alignment
The fragment current position is the relative (counted from the
fragment start) position of the currently shown left end of the
respective fragment. A dot (•) is shown to the left of the position
if the fragment is completely to the left of currently displayed
area.
3. Exit ContigExpress
Now you have finished the ContigExpress tutorial.
Close ContigExpress as you would close any other Windows
application.
- 62 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 63

Vector NTI User Manual
第九章 Vector NTI Tools
一. Introduction
Vector NTI suiteII 將搜尋介面 Entrez/PubMed search 整合至整個
分析工具中,透過網際網路的連結,直接與 NCBI 資料庫進行串連。
注意:此等為附加功能非原廠主要提供之功能,後端連結 NCBI
資料庫的適用性,可能會因 NCBI 的更動而有所影響,原廠將以提供
更動時最新版本的連結更新程式。
二. PubMed/Entrez Search
此等工具是藉由操作介面直接與 NCBI 資料庫做連結,資料庫的
更新跟隨 NCBI 而更新。使用者只需下載所需資料,並可存放
於”Database Explore”中;另外提供一個文獻瀏覽器(Citation viewer)
可進行文獻瀏覽。
開始Vector NTI Suite 7PubMed/Entrez Search (
)
i
Database 選擇可分為
Citation-文獻
- 63 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 64

Vector NTI User Manual
Nucleotide- 核酸序列
Protein- 氨基酸序列
Structure- 蛋白質 3-D 結構序列與圖形
NOTES: 選擇搜尋方式可以由輸入欄後方的選項捲軸分類。.
若是要利用多重因子進行搜尋,在各個因子分別輸入時不可以
按”ENTER”,因為此時會直接進行搜尋,要輸入下一個因子時,只要
按”SPACE”即可。
多重因子搜尋時,可利用各因子之間 Or, And or Not 的關係,進
行交叉搜尋。
Defining Search Conditions
Before you initiate the search, select EditSettings, opening the
Settings dialog box.
搜尋完後可將結果 Check the Save Results… 來存檔。
The History buffer contains the last queries and query results. You
can move through the history by using Back and Forward buttons.
Internet 設定以使用者網路設定為主,因地制宜。.
In the Publication Date section, enter publication dates to narrow the
search to a certain date range. In the Entrez date section, enter acceptable
molecule submission dates.
Click OK to return to the PubMed/Entrez Search dialog box.
When you have entered all search conditions, press the Search
- 64 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 65

Vector NTI User Manual
button ( ). The search results are displayed in the list pane in the
bottom half of the dialog box.
Reviewing Search Results
Article or molecule summaries are listed in the Results Pane. There
are several techniques to download the citation or molecule results:
Double-click on its identification code for the selected object or
select multiple objects
Select Open DNA and Protein 將直接於Vector NTI 視窗中呈現;而
文獻的”Abstract”部分將呈現於”Citation Viewer”.
Select Export from the shortcut menu to download the selected item
and places it in a file you specify.
Drag and drop selected object(s) from the Results Pane into
Database Explorer or Windows Explorer folders.
若是將所要呈現的資料多重選取,再選擇開啟;此時將會於相對
應的功能視窗中呈現出來。.
Saving Search Results
假使使用者選用 Save As 此時將是只存取搜尋結果的畫面;而非
各筆資料。若要將資料下載至電腦中可將所要的資料選取後,至視窗
上方的”Search” Result Open/ Save to…中,此時會出現如下對
話方塊
- 65 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 66

Vector NTI User Manual
此時若選擇”Import”資料將會直接進入”Database Explore”相對應
的資料夾中;若是選”Open”則會將所有資料以多重視窗呈現。
三. Citation Viewer
Vector NTI suite II 提供的 Citation Viewer 只針對文獻中
的”Abstract”部分,資料的可由”Database Explore”中的 Citation 資料
夾,或是由 PubMed/Entrez Search 結果輸入。
To open Citation Viewer, select Citation Viewer in the program
- 66 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 67

Vector NTI User Manual
group or folder where you installed Vector NTI. If opened from
Database Explorer, Citation Viewer is empty and must be loaded from
other sources, listed below:
From the PubMed/Entrez Search results, or from the Citations Table
in Database Explorer, drag and drop citations into Citation Viewer. It
helps if all of your screens are in “Partial Screen Size” to do this. If you
transfer multiple files, all of the abstracts will open in Citation Viewer.
You can alternate between references by pressing the Back and Forward
buttons on the toolbar (they resemble the Back and Forward buttons on
your Web browser) or you can select from the list of open citations under
the Windows menu option.
Opening Citation Viewer
You can open an individual citation into the Citations Viewer
window from the PubMed/Entrez Search display window or from the
Citations Table in Database Explorer. Simply double-click the entry in
either location to load Citations Viewer.
Citation viewer registers its file types, so doubleclicking on
some .ca6, .cq or .ct file in Windows Explorer will cause Citation Viewer
to run and open that file.
Elements of Citation Viewer
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
- 67 -
Page 68

Vector NTI User Manual
The Citations Viewer display window shares many of the same
features found in other Vector NTI Suite applications:
The window title bar displays the name of the open object.
Click the maximize box (
) in the upper right corner on the title
bar to maximize the window, filling the entire Vector NTI workspace.
A display window is divided into two panes: a Text Pane containing
folders with descriptions of the citation such as author, source, and public
database identification data. The Abstract Pane displays the abstract for
the citation.
A vertical, movable split bar divides the two panes.
All Vector NTI Suite windows have a Main Menu and toolbars
where many different options and operations can be initiated. Most of
the toolbar buttons in Citation Viewer are similar to those you have seen
in other display windows. All Vector NTI Suite toolbar buttons are
summarized in Chapter ss of this manual. The toolbar buttons you will
use in this section are displayed when needed.
Back and forward buttons will bring up the previous or next viewed
document if it is still open.
Note: If the citation contains an URL to its full text or abstract in
the Internet, that URL will be displayed in Text Pane. Clicking on it
causes the default Internet browser to open to the appointed location.
The following feature can be performed in Citation Viewer.
Copying selected data to the clipboard
Exporting data to external programs
Printing data
Modifying the Citation Viewer layout
Saving data
- 68 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 69

Vector NTI User Manual
如何執行反安裝程式
1. 開始→程式集→Vector NTI suite→License Manager 此時會
出現一個管理視窗:
點選
- 69 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
Page 70

Vector NTI User Manual
此時在”Key”的空格內點鍵入”UNREGISTER”
之後再以滑鼠指標按下”Apply”鍵,此時會出現另一個視窗告知
有另一組 Unregister key,請記下這組號碼通知本公司即可。
- 70 -
源資國際生物科技股份有限公司 Copyright 2003 All right reserved
 Loading...
Loading...